-
近年来,随着广谱抗生素的使用、艾滋病病毒感染、肿瘤放疗/化疗以及器官移植患者的不断增多,导管插管等越来越多的生物材料应用于人体,侵袭性真菌感染的发病率显著上升,对人类健康乃至生命造成严重威胁。在临床真菌感染中,白假丝酵母(Candida albicans)是最常见的致病真菌之一[1]。研究表明,白假丝酵母能通过黏附于人上皮及植入的导管或支架等表面,形成生物被膜(biofilm),从而在免疫功能低下的人群中导致系统性感染。白假丝酵母生物被膜的一个严重后果是对临床常用抗真菌药物呈高度耐药。与浮游型白假丝酵母相比,生物被膜型白假丝酵母对两性霉素B、氟康唑的敏感性仅是浮游菌的几十分之一。生物被膜形成是导致临床上许多系统性、反复性感染的重要因素,是抗真菌感染治疗失败的主要原因之一[2-3]。因此,研究开发抗白假丝酵母生物被膜药物对于抗真菌感染的治疗具有重要意义。
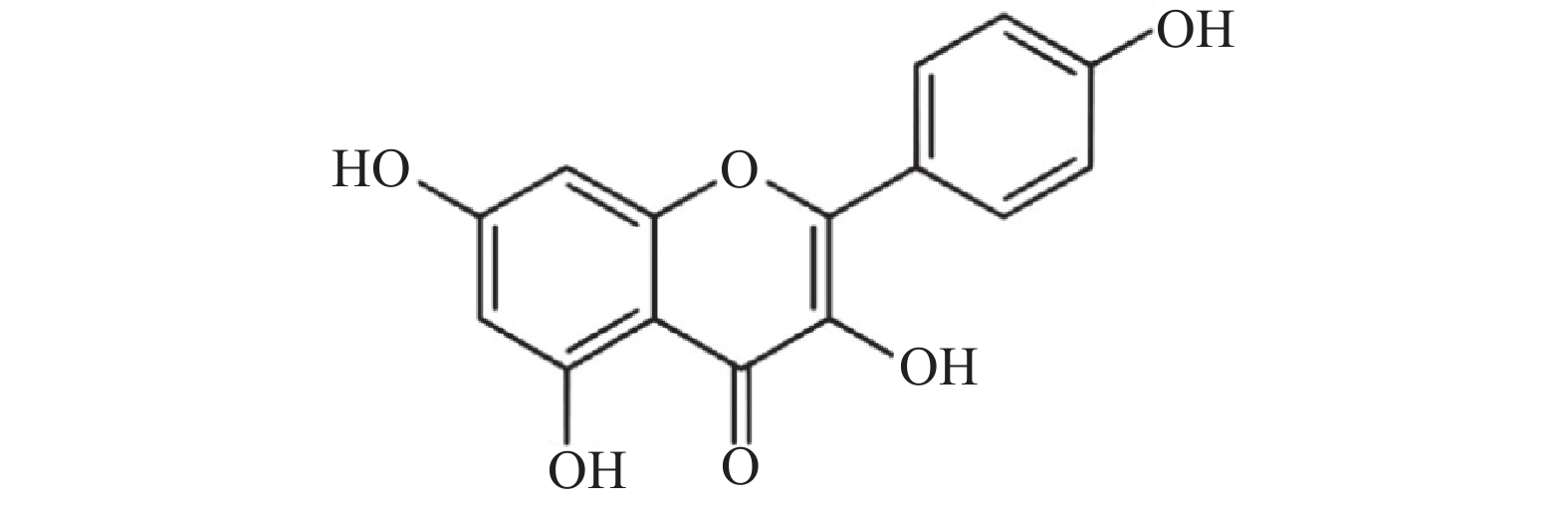
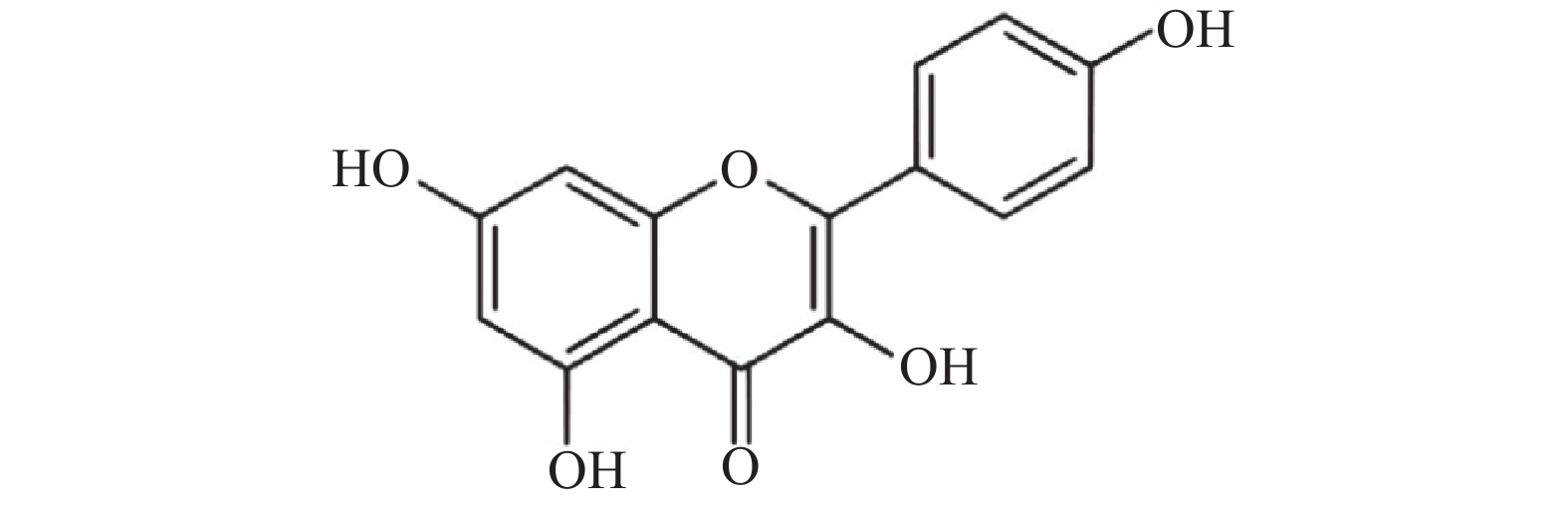
山奈酚(kaempferol,KAE)又名山柰素、山柰黄酮醇,其分子结构见图1。山奈酚属于黄酮类化合物,主要来源于姜科植物山柰的根茎。同时,该化合物广泛存在于多种蔬菜及水果中。研究显示,山奈酚具有抗肿瘤、抗炎、抗氧化、抑制血小板聚集及抗病毒等多种生物学活性[4-6]。本文旨在研究山奈酚抗白假丝酵母生物被膜活性并探索其潜在作用机制,为临床抗真菌感染提供新思路。
-
白假丝酵母国际通用株SC5314(C. albicans SC5314)由上海市皮肤病医院中心实验室保存。山奈酚(美国Sigma公司),二甲亚砜(DMSO,国药化学试剂有限公司)。将山奈酚溶于DMSO配制成母液,使用时以RPMI1640稀释至所需浓度。XTT(化学名:2,3-bis(2-methoxy-4-nitro-5-sulfo-phenyl)-2H-tetrazolium-5-carboxanilide)和甲萘醌试剂(美国Sigma公司),分别用PBS、丙酮溶解配制成母液。真菌RNA抽提试剂盒(北京天恩泽基因科技公司),RNA反转录、PrimeScript RT Master Mix Perfect RealTime及SYBR Premix ExTaqTM试剂(TaKaRa生物公司)。
沙氏固体培养基(SDA):蛋白胨10 g, D-葡萄糖40 g,琼脂粉20 g,以去离子水溶解并定容至1 000 ml,高压灭菌(121 ℃,15 min),室温冷却凝固后备用。YPD液体培养基:蛋白陈10 g,酵母提取物10 g,D-葡萄糖20 g,以去离子水溶解并定容至1 000 ml,分装后高压灭菌(121 ℃,15 min)备用。RPMI1640液体培养基:将RPMI1640粉末(Gibco BRL公司)10 g,MOPS(涯泰生物科技公司)34.5 g,NaHCO32.0 g,以去离子水溶解并定容至1 000 ml,用NaOH调节pH至7.0,定容至1 000 ml,微孔滤膜过滤除菌,4 ℃保存。
-
恒温振荡培养箱(江苏太仓市实验设备厂),高速冷冻离心机(德国Eppendorf公司),超净化工作台(苏州安泰空气技术有限公司),96孔细胞培养板(美国Corning公司),Infinite M200多功能酶标仪(Austria TECAN公司),ABl7500实时定量 RT-PCR仪(Applied Biosystems公司)。
-
挑取白假丝酵母SC5314甘油冻存菌划于SDA平板,置于30 ℃恒温培养箱培养3 d,待长出单克隆后,挑取菌株单克隆接种于新鲜YPD液体培养基中,再于30 ℃、200 r/min振荡培养过夜(16 h),使其到达对数生长期后期。
-
白假丝酵母生物被膜培养实验参照文献[7]进行。离心收集上述培养基至对数生长后期的白假丝酵母菌液,PBS洗涤3次,重悬于RPMI1640培养基,计数并调节菌液浓度为1×106 CFU/ml。在96孔细胞培养板每孔加入100 μl上述菌液,37 ℃静置培养2 h,弃上清液,于各孔中分别加入100 μl含有不同浓度山奈酚的RPMIl640培养基,37 ℃继续静置培养24 h。
-
离心收集培养至对数生长后期的白假丝酵母菌液,PBS洗涤3次,重悬于RPMI1640培养基,计数并调节菌液浓度为1×106 CFU/ml。在96孔细胞培养板每孔加入100 μl上述菌液,37 ℃静置培养2 h,弃上清液,于各孔中加入新鲜RPMIl640培养基,37 ℃继续静置培养24 h,弃上清液,PBS洗涤2次,于各孔中分别加入100 μl含有不同浓度山奈酚的RPMIl640培养基,37 ℃继续静置培养24 h。
-
取出上述培养的生物被膜,弃上清液,PBS洗2次,随后加入200 μlXTT-甲萘醌溶液(含有0.5 mg/mlXTT-1 μmol/L甲萘醌),于37 ℃黑暗处静置孵育2 h后取出。采用多功能酶标检测仪于492 nm处测定光密度(OD)值。
-
生物被膜基质(biomass)含量测定参照文献[8]方法进行。在预先放置有硅胶片(1.5 cm×1.5 cm,美国Bentec医药公司)的12孔培养板中,每孔加入2 ml白假丝酵母菌液(1×106 CFU/ml于RPMI1640培养基中),黏附2 h,弃上清液,加入含有不同浓度山奈酚的新鲜RPMI1640培养基,37 ℃继续静置培养24 h,弃上清液,PBS洗涤2次,于各孔中分别加入100 μl含有不同浓度山奈酚的RPMIl640培养基,37 ℃继续静置培养24 h,取出上述培养的生物被膜,弃上清液,PBS洗2次,晾干至恒重并称重,所得重量减去硅胶片本身质量即为生物被膜基质量。
-
将过夜培养至对数生长后期的白假丝酵母,于次日按照1%接种于新鲜YPD液体培养基,30 ℃继续振荡培养4 h,PBS洗涤2次,重悬于含有16 μg/ml的山奈酚YPD+FBS培养基(含有10%胎牛血清),调整菌浓度为l×l06 CFU/ml,于细胞培养板中37 ℃静置培养3.5 h,显微镜下观察白假丝酵母菌丝形成情况。
-
采用水-烃两相分离法测定细胞表面疏水性[9]。将过夜培养至对数生长后期的白假丝酵母于次日按照1%量接种于新鲜YPD液体培养基,30 ℃继续振荡培养4 h,随后加入不同浓度山奈酚,于30 ℃继续振荡培养4 h,离心收集菌液以PBS洗涤2次,重悬于YPD液体培养基,调整菌液至OD600=1.0,每组取1.2 ml菌悬液于另一离心管中,加入0.3 ml正辛烷,涡旋振荡混匀3 min,室温静置使两相分离,立即测定上层水相的OD600值,以未加正辛烷的YPD培养基为阴性对照。白假丝酵母细胞表面疏水性值的计算公式:相对细胞表面疏水性=[(OD600对照组−OD600实验组)/OD600对照组]×100%。
-
应用Primer Premier5软件设计用于实时定量RT-PCR扩增的目的基因引物序列(表1)。引物由上海生工生物工程技术有限公司合成。
表 1 引物序列
引物序列(5′-3′) HWP1-F TGGTGCTATTACTATTCCGG HWP1-R CAATAATAGCAGCACCGAAG EFG1-F TATGCCCCAGCAAACAACTG EFG1-R TTGTTGTCCTGCTGTCTGTC CPH1-F ATGCAACACTATTTATACCTC CPH1-R ATGCAACACTATTTATACCTC ALS1-F TTGGGTTGGTCCTTAGATGG ALS1-R ATGATTCAAAGCGTCGTTC ALS3-F CTAATGCTGCTACGTATAATT ALS3-R CCTGAAATTGACATGTAGCA CSH1-F CTGTCGGTACTATGAGATTG CSH1-R GATGAATAAACCCAACAACT TUP1-F GATTGACGAG TCCTCCAACG TUP1-R AAACCAACCTATCGCCATCA NRG1-F TATCAGTATG CTGCTCCTCC NRG1-R GGAGTTGGCCAGTAAATCAC BCR1-F AGTATAATGCTCCTGGTAAGAA BCR1-R ACGTAAAGGAGGCACGGCATA 18S rRNA-F AATTACCCAATCCCGACAC 18S rRNA-R TGCAACAACTTTAATATACGC -
将培养至对数生长后期的白假丝酵母菌液,PBS洗涤3次,重悬于RPMI1640培养基,计数并调节菌液浓度为l×l06 CFU/ml。将上述菌液在细胞培养瓶中37 ℃静置培养2 h,弃上清液,随后加入含有16 μg/ml山奈酚的新鲜RPMIl640培养基,37 ℃继续静置培养24 h。离心收集菌体,PBS洗涤3次,按照北京天恩泽基因科技公司真菌RNA抽提试剂盒的操作说明进行总RNA的抽提,抽提完毕后加入100 μl去除RNA酶的水(经DEPC处理)溶解RNA,采用分光光度计测定RNA纯度及含量,A260/A280比值在1.8~2.0之间为合格。按照TaKaRa生物公司的反转录试剂盒操作说明,将上述RNA反转录为cDNA。取上述逆转录产物进行PCR扩增,以18S rRNA作为内参基因。反应条件为预变性95 ℃,30 s,重复40个循环。循环参数为:95 ℃,5 s;60 ℃,20 s;72 ℃,30 s。溶解曲线采用60~95 ℃,温度改变速率为每秒0.1 ℃。扩增产物采用ABI 7500 SDS软件系统进行分析。采用2−(⊿⊿Ct)法表示基因表达水平。
-
实验数据应用GraphPad Prism 6.0软件进行作图及统计学检验,以(
$\bar x \pm s$ )表示,每个实验至少重复3次,以P<0.05为差异显著,P<0.01为差异极显著。 -
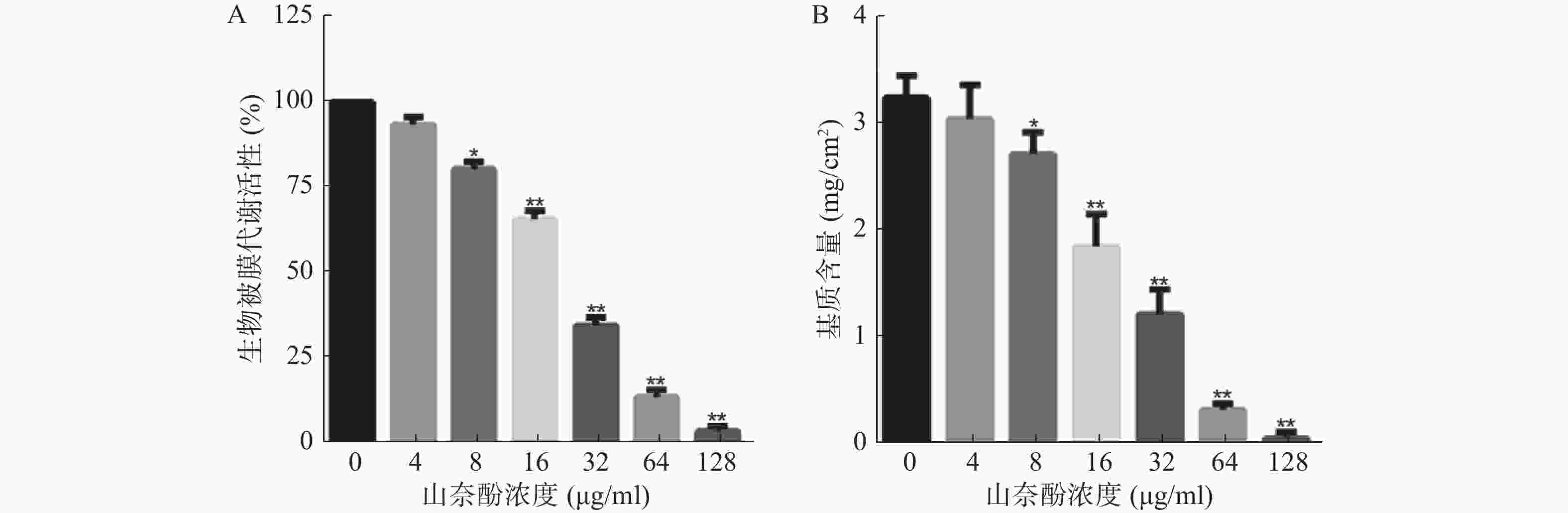
以不同浓度的山奈酚作用于白假丝酵母生物被膜形成早期(黏附2 h),于37 ℃继续培养24 h后测定生物被膜代谢活性。XTT测定结果显示:山奈酚可抑制白假丝酵母生物被膜形成,且呈剂量依赖性。当山奈酚浓度为8 μg/ml时,生物被膜形成被明显抑制。当32 μg/ml的山奈酚作用于生物被膜时,其代谢活性约为对照组的35%,经128 μg/ml的山奈酚处理的白假丝酵母几乎不能形成生物被膜(图2A)。
由于生物被膜基质含量是显示白假丝酵母生物被膜形成能力的一个重要特征,我们进一步考察了山奈酚对生物被膜基质含量的影响。结果表明:与对照组相比,经山奈酚处理的生物被膜基质含量显著下降,呈现剂量依赖性。当32 μg/ml的山奈酚作用于白假丝酵母生物被膜时,其基质产生量不足对照组的50%(图2B)。
-
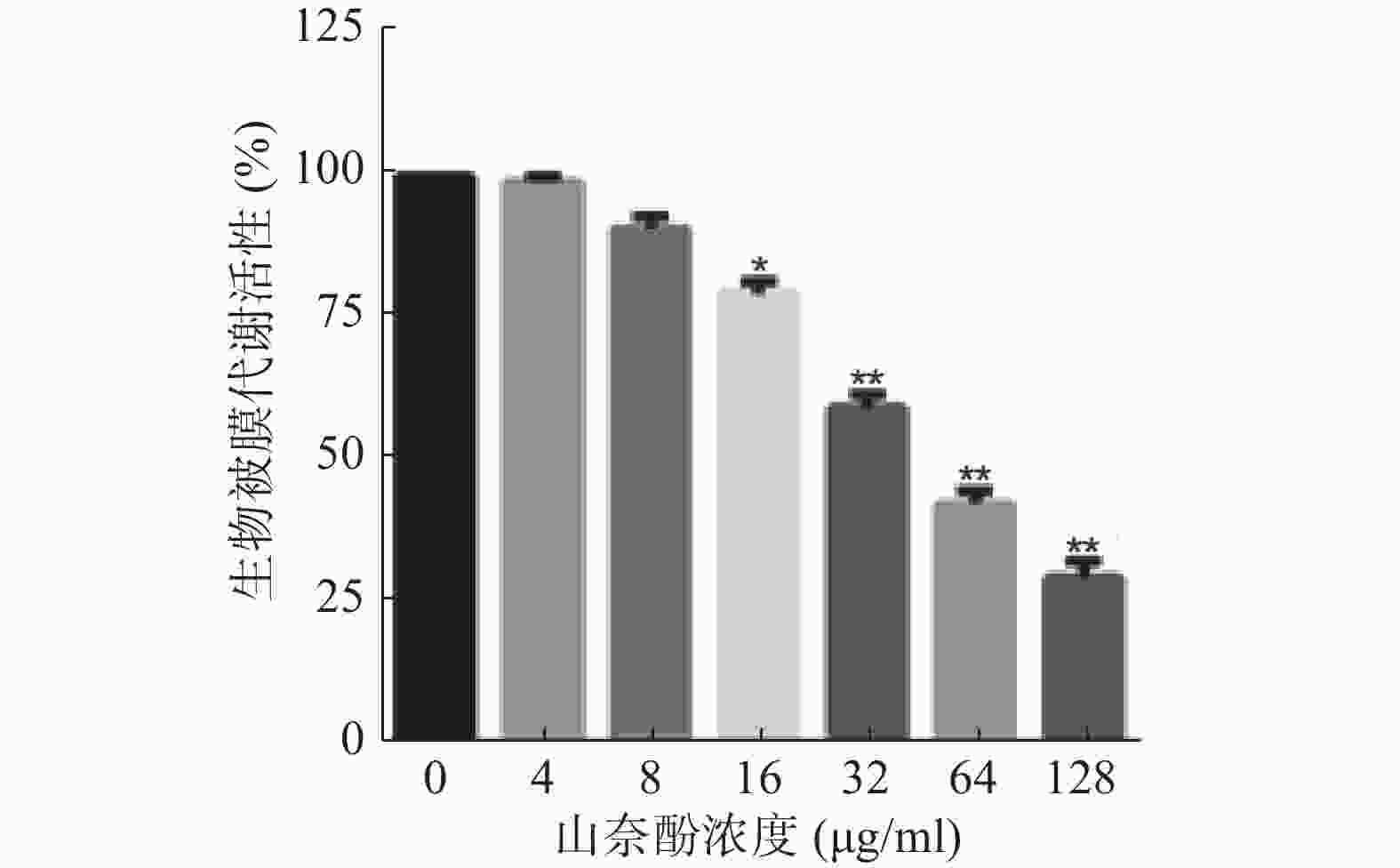
将不同浓度的山奈酚作用于成熟白假丝酵母生物被膜(培养24 h),于37 ℃继续培养24 h后测定生物被膜代谢活性。结果显示:16 μg/ml的山奈酚具有明显的抗成熟生物被膜活性。当32 μg/ml的山奈酚作用于成熟生物被膜时,其代谢活性约为对照组的60%,经128 μg/ml的山奈酚处理的白假丝酵母生物被膜活性约为对照组的28%(图3)。
-
由于白假丝酵母菌丝形成能力与生物被膜密切相关,因此,本实验进一步考察山奈酚对菌丝形成的影响。未加药的对照组白假丝酵母在含有10%胎牛血清的YPD培养基中可以形成正常菌丝,而当16 μg/ml的山奈酚作用于白假丝酵母时,菌丝生长受到明显抑制,主要以酵母型菌生长(图4)。
-
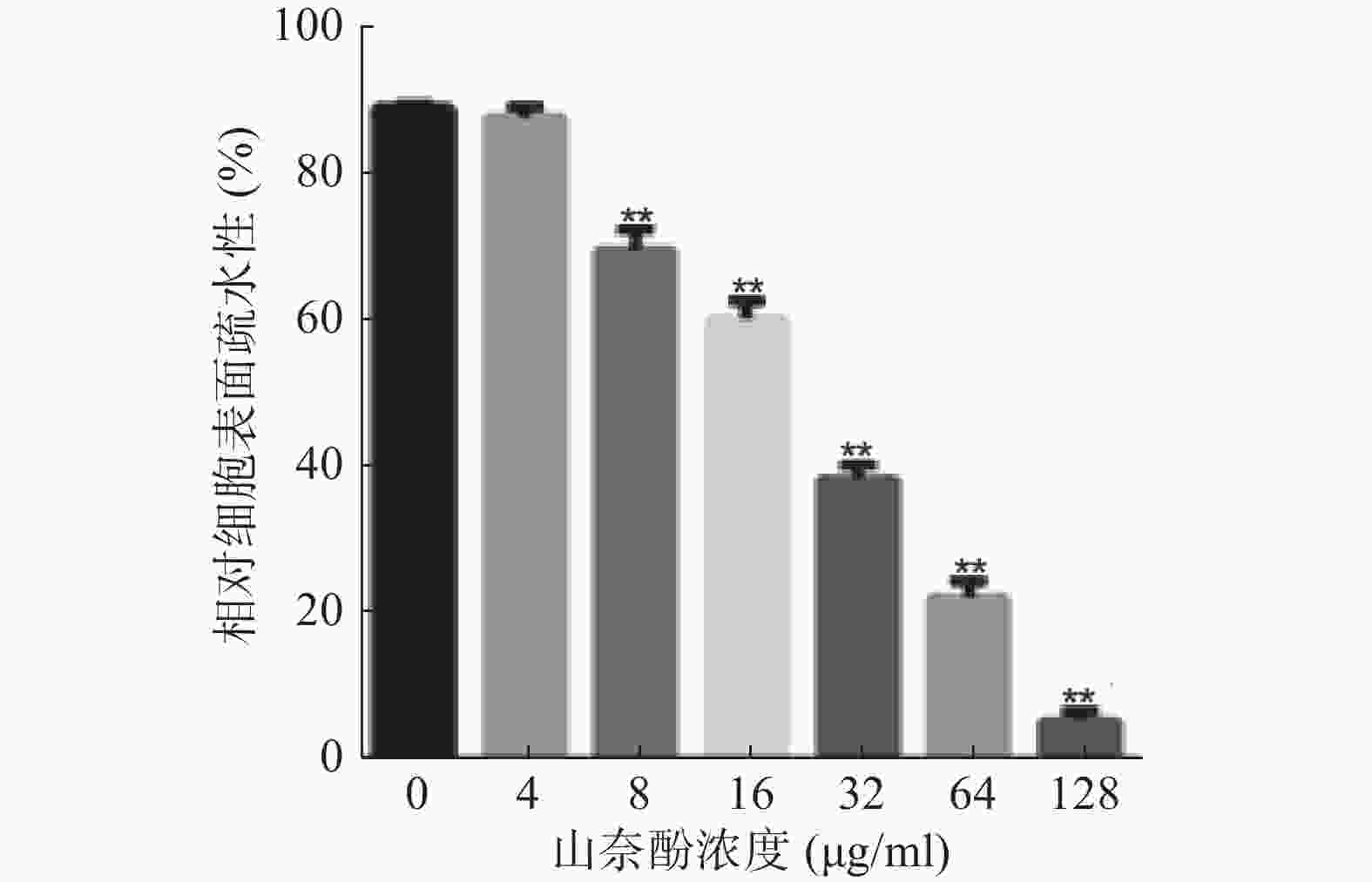
黏附是白假丝酵母生物被膜形成的早期关键步骤,而细胞表面疏水性对黏附具有重要影响,因此,本实验考察山奈酚对白假丝酵母细胞表面疏水性的影响。与对照组相比,4 μg/ml的山奈酚对白假丝酵母细胞表面疏水性无明显影响,而当山奈酚的作用浓度为8 μg/ml时,其细胞表面疏水性明显下降,且呈现剂量依赖性(图5)。
-
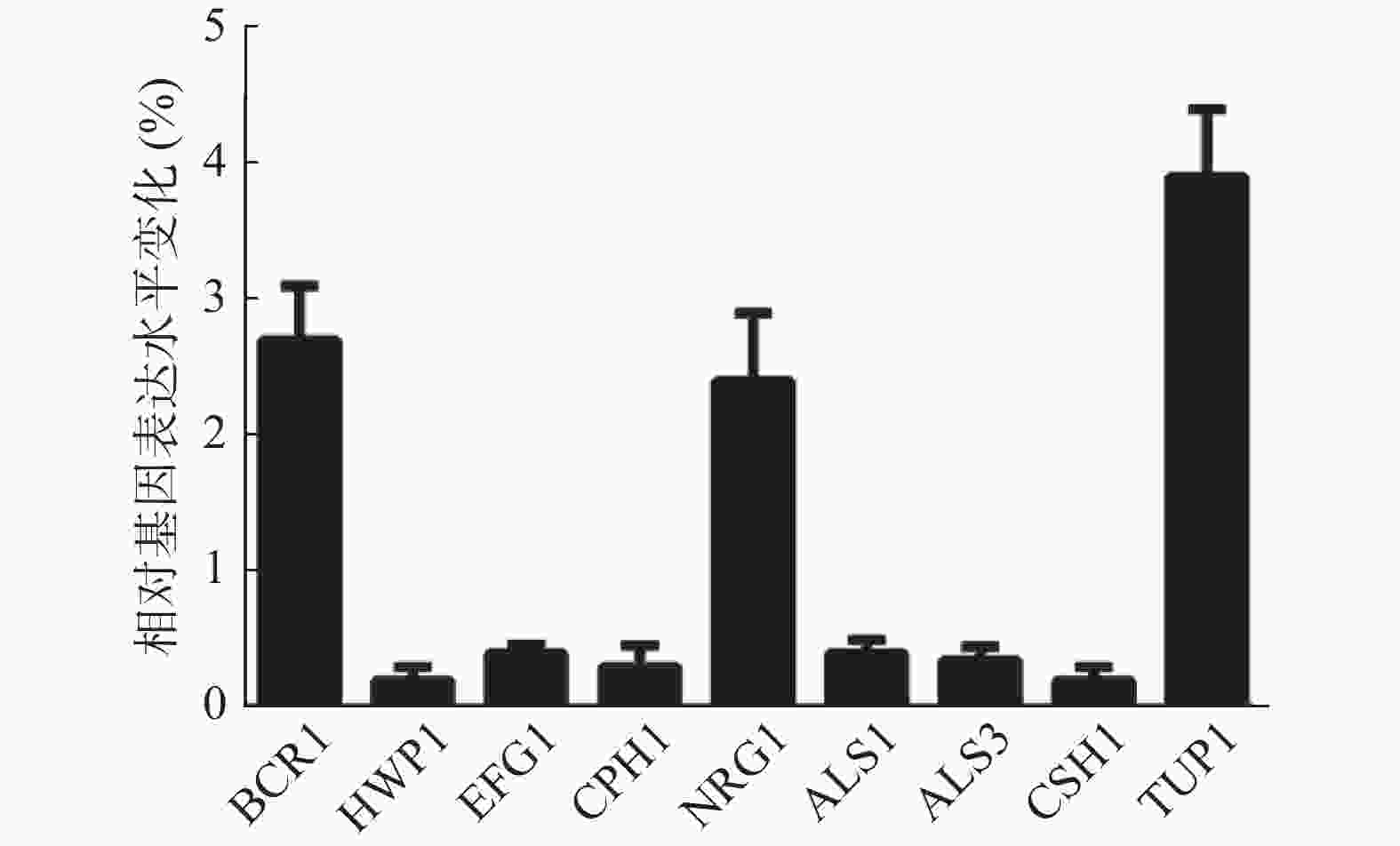
由于山奈酚可以影响白假丝酵母菌丝形成及细胞表面疏水性,因此,本实验采用实时定量RT-PCR法测定山奈酚对白假丝酵母菌丝形成及细胞表面疏水性相关基因表达的影响。如图6所示,经16 μg/ml山奈酚处理的白假丝酵母生物被膜中菌丝形成相关基因BCR1、NRG1和TUP1的基因表达分别升高了2.7、2.4和3.9倍,HWP1、EFG1和CPH1基因表达显著下降,同时,细胞表面疏水性相关基因ALS1、ALS3和CSH1的表达明显下降。
-
近年来,随着免疫低下人群的不断增多以及越来越多的医疗器材应用于人体,真菌感染率大幅增加,其中,以生物被膜型白假丝酵母感染较为常见,常导致反复性感染及耐药,是临床抗感染治疗的一个重要难题。因此,寻找开发具有抗生物被膜活性的化合物对于防治真菌感染具有重要意义。
天然化合物作为药物的一个重要组成部分,被广泛应用于各种疾病的治疗。山奈酚是最常见的膳食类黄酮化合物之一,广泛存在于花菜、茶叶及柚子等多种植物中,具有抗炎、抗氧化及抗肿瘤等多种生物学活性。Shao等报道山奈酚具有抗浮游型真菌活性并抑制耐药相关外排泵基因的表达[10]。本实验结果显示,山奈酚具有抗白假丝酵母生物被膜活性,不仅可以抑制生物被膜形成,对于成熟生物被膜也有抑制作用。研究表明,白假丝酵母生物被膜基质中富含碳水化合物及DNA,可以通过形成物理屏障阻碍抗真菌药物进入胞内,因此,其含量与耐药程度密切相关。有报道称,生物被膜基质可以隔离放射性标记的氟康唑,将两性霉素B作用于去除基质的念珠菌生物被膜,其抗菌活性与为去除基质的对照组相比明显增强[11-13]。在本实验中,经山奈酚处理的白假丝酵母生物被膜基质水平明显下降,这可能是该化合物发挥抗生物被膜活性的机制之一。
白假丝酵母生物被膜主要由3部分构成,即基底芽生孢子层、菌丝成分以及细胞外多聚基质,其中,菌丝成分是构成完整生物被膜的重要元件,酵母态到菌丝态的转换是其形成生物被膜的关键。本实验结果显示,山奈酚可以明显抑制白假丝酵母菌丝形成。进一步的实时定量RT-PCR测定结果表明,山奈酚对菌丝形成相关基因的表达具有调控作用。HWP1是菌丝特异性表达基因,该基因缺失型生物被膜极易从附着物上脱落[14]。山奈酚对生物被膜的抑制作用可能与其下调HWP1基因表达有关。同时,经山奈酚处理的白假丝酵母中2个调控菌丝形成信号通路的重要转录因子EFG1和CPH1的表达也明显下降。TUP1编码的转录因子对菌丝形成具有抑制作用,可抑制菌丝形成相关基因的表达。有研究报道,TUP1敲除型白假丝酵母即使在适于酵母菌生长条件下也表现为菌丝过度生长[15]。在白假丝酵母菌丝及生物被膜形成抑制剂farnesol处理的白色念珠菌中,TUP1基因呈过度表达[7]。NRG1是另一菌丝生长抑制型转录因子,在菌丝诱导条件下其mRNA和蛋白表达水平均下降,该过程依赖cAMP/PKA信号通路。NRG1对菌丝形成具有极强的抑制作用,可与TUP1共同抑制菌丝形成[16-18]。本实验中,TUP1和NRG1在山奈酚处理组中呈现高表达与菌丝形成抑制的生物学表型相一致。
白假丝酵母生物被膜的形成分为起始黏附、微克隆形成以及生物被膜成熟3个阶段,其中,细胞表面疏水性与起始黏附密切相关,对于正常生物被膜的形成至关重要[19]。本实验结果显示,与对照组相比,经山奈酚处理的白假丝酵母细胞表面疏水性明显下降。与此相一致的是,细胞表面疏水性相关基因CSH1的表达在山奈酚处理组中明显下降。已有研究表明,ALS家族基因在促进白假丝酵母黏附及生物被膜的形成中发挥重要作用[20]。本实验中,该家族中的ALS1和ALS3基因在山奈酚处理组中表达下降。文献报道转录因子BCR1位于ALS1和ALS3基因上游,参与调控白假丝酵母的黏附及生物被膜形成。与ALS1和ALS3基因下调相反,本实验中经山奈酚处理的白假丝酵母中BCR1基因表达升高,提示山奈酚可能通过不依赖于BCR1的其他信号通路来调控ALS1和ALS3基因的表达[21]。综上所述,山奈酚可能通过抑制白假丝酵母的黏附和菌丝形成从而发挥抗生物被膜作用。
综上所述,本研究首次揭示了山奈酚的体外抗白假丝酵母生物被膜活性,其作用机制可能与抑制白假丝酵母菌丝形成和降低其细胞表面疏水性相关,这为治疗临床真菌生物被膜相关感染提供了新思路。
Study on the antibiofilm activity of kaempferol in Candida albicans
-
摘要:
目的 研究山奈酚抗白假丝酵母生物被膜的作用及其可能机制。 方法 测定山奈酚对白假丝酵母处于形成过程中的生物被膜和成熟生物被膜代谢活性的影响;测定山奈酚对生物被膜基质产生水平的影响;显微镜下观察山奈酚对菌丝形成的抑制作用;水-烃两相分离法测定山奈酚对白假丝酵母细胞表面疏水性的影响;实时定量RT-PCR法测定山奈酚对生物被膜形成相关基因表达的影响。 结果 山奈酚抑制白假丝酵母生物被膜形成,且呈剂量依赖性,同时具有抗成熟生物被膜作用,显著降低生物被膜基质含量;与对照组相比,山奈酚明显抑制白假丝酵母菌丝形成并降低其细胞表面疏水性;经山奈酚处理的白假丝酵母生物被膜形成相关基因BCR1、NRG1和TUP1的表达升高,同时HWP1、EFG1、CPH1、ALS1、ALS3和CSH1的表达下降。 结论 山奈酚具有抗白假丝酵母生物被膜活性,其机制与抑制菌丝形成及降低其细胞表面疏水性相关。 Abstract:Objective To study the action of kaempferol (KAE) against Candida albicans biofilms and explore the potential mechanisms. Methods Biofilm metabolic activity assay was used to investigate the action of KAE against C. albicans biofilm formation as well as mature biofilm. The inhibition of KAE in hyphal formation was examined by microscope. The water-hydrocarbon two-phase separation assay was used to test the effect of KAE on the cell surface hydrophobicity of C. albicans. The mRNA expression of the genes involved in biofilm formation was determined by real time RT-PCR. Results KAE showed inhibition effect on C. albicans biofilm formation in a dose-dependent manner. Moreover, KAE inhibited mature biofilm. The biomass of biofilm was reduced upon KAE treatment. KAE inhibited hyphal formation and reduced the cell surface hydrophobicity of C. albicans. In the presence of KAE, the mRNA expression of the genes involved in biofilm formation was changed, with the up-regulation of BCR1,NRG1,TUP1 and down-regulation of HWP1,EFG1,CPH1,ALS1,ALS3 and CSH1. Conclusion KAE showed antifungal activity against C. albicans biofilm. The mechanisms may relate to the inhibition of hyphal formation and reduction of cell surface hydrophobicity. -
Key words:
- kaempferol /
- Candida albicans /
- biofilm /
- adhesion /
- hyphae
-
表 1 引物序列
引物序列(5′-3′) HWP1-F TGGTGCTATTACTATTCCGG HWP1-R CAATAATAGCAGCACCGAAG EFG1-F TATGCCCCAGCAAACAACTG EFG1-R TTGTTGTCCTGCTGTCTGTC CPH1-F ATGCAACACTATTTATACCTC CPH1-R ATGCAACACTATTTATACCTC ALS1-F TTGGGTTGGTCCTTAGATGG ALS1-R ATGATTCAAAGCGTCGTTC ALS3-F CTAATGCTGCTACGTATAATT ALS3-R CCTGAAATTGACATGTAGCA CSH1-F CTGTCGGTACTATGAGATTG CSH1-R GATGAATAAACCCAACAACT TUP1-F GATTGACGAG TCCTCCAACG TUP1-R AAACCAACCTATCGCCATCA NRG1-F TATCAGTATG CTGCTCCTCC NRG1-R GGAGTTGGCCAGTAAATCAC BCR1-F AGTATAATGCTCCTGGTAAGAA BCR1-R ACGTAAAGGAGGCACGGCATA 18S rRNA-F AATTACCCAATCCCGACAC 18S rRNA-R TGCAACAACTTTAATATACGC -
[1] ACHKAR J M, FRIES B C. <italic>Candida</italic> infections of the genitourinary tract[J]. ClinMicrobiolRev,2010,23(2):253-273. [2] ANDERSON J B. Evolution of antifungal-drug resistance: mechanisms and pathogen fitness[J]. Nat Rev Microbiol,2005,3(7):547-556. [3] GULATI M, NOBILE C J. <italic>Candidaalbicans</italic> biofilms: development, regulation, and molecular mechanisms[J]. Microbes Infect,2016,18(5):310-321. doi: 10.1016/j.micinf.2016.01.002 [4] 陈育华, 周克元, 袁汉尧. 山奈酚药效的研究进展[J]. 广东医学, 2010, 31(8):1064-1066. doi: 10.3969/j.issn.1001-9448.2010.08.058 [5] KIM B W, LEE E R, MIN H M, et al. Sustained ERK activation is involved in the kaempferol-induced apoptosis of breast cancer cells and is more evident under 3-D culture condition[J]. Cancer Biol Ther,2008,7(7):1080-1089. [6] RAJENDRAN P, RENGARAJAN T, NANDAKUMAR N, et al. Kaempferol, a potential cytostatic and cure for inflammatory disorders[J]. Eur J Med Chem,2014,86:103-112. [7] CAO Y Y, CAO Y B, XU Z, et al. cDNA microarray analysis of differential gene expression in <italic>Candidaalbicans</italic> biofilm exposed to farnesol[J]. Antimicrob Agents Chemother,2005,49(2):584-589. doi: 10.1128/AAC.49.2.584-589.2005 [8] NOBILE C J, ANDES D R, NETT J E, et al. Critical role of Bcr1-dependent adhesins in C. albicans biofilm formation <italic>in vitro</italic> and <italic>in vivo</italic>[J]. PLoS Pathog,2006,2(7):e63. [9] YAN Y, TA NF, MIAO H, et al. Effect of shikoninagainst <italic>Candida albicans</italic>biofilms[J]. Front Microbiol,2019,10:1085. doi: 10.3389/fmicb.2019.01085 [10] SHAO J, ZHANG M X, WANG T M, et al. The roles of CDR1, CDR2, and MDR1 in kaempferol-induced suppression with fluconazole-resistant <italic>Candidaalbicans</italic>[J]. Pharm Biol,2016,54(6):984-992. doi: 10.3109/13880209.2015.1091483 [11] SUN F J, QU F, LING Y, et al. Biofilm-associated infections: antibiotic resistance and novel therapeutic strategies[J]. Future Microbiol,2013,8(7):877-886. doi: 10.2217/fmb.13.58 [12] AL-FATTANI M A, DOUGLAS L J. Biofilm matrix of <italic>Candidaalbicans</italic> and <italic>Candidatropicalis</italic>: chemical composition and role in drug resistance[J]. J Med Microbiol,2006,55(Pt 8):999-1008. [13] NETT J, LINCOLN L, MARCHILLO K, et al. Putative role of beta-1, 3 glucans in <italic>Candidaalbicans</italic> biofilm resistance[J]. Antimicrob Agents Chemother,2007,51(2):510-520. doi: 10.1128/AAC.01056-06 [14] CHAFFIN W L. <italic>Candidaalbicans</italic> cell wall proteins[J]. Microbiol Mol Biol Rev,2008,72(3):495-544. doi: 10.1128/MMBR.00032-07 [15] BRAUN B R, JOHNSON A D. Control of filament formation in <italic>Candidaalbicans</italic> by the transcriptional repressor TUP1[J]. Science,1997,277(5322):105-109. doi: 10.1126/science.277.5322.105 [16] BRAUN B R, KADOSH D, JOHNSON A D. NRG1, a repressor of filamentous growth in <italic>C.albicans</italic>, is down-regulated during filament induction[J]. EMBO J,2001,20(17):4753-4761. doi: 10.1093/emboj/20.17.4753 [17] CLEARY I A, MULABAGAL P, REINHARD S M, et al. Pseudohyphal regulation by the transcription factor Rfg1p in <italic>Candidaalbicans</italic>[J]. Eukaryotic Cell,2010,9(9):1363-1373. doi: 10.1128/EC.00088-10 [18] LU Y, SU C, WANG A, et al. Hyphal development in <italic>Candidaalbicans</italic> requires two temporally linked changes in promoter chromatin for initiation and maintenance[J]. PLoS Biol,2011,9(7):e1001105. doi: 10.1371/journal.pbio.1001105 [19] SENEVIRATNE C J, JIN L, SAMARANAYAKE L P. Biofilm lifestyle of <italic>C.andida</italic>: a mini review[J]. Oral Dis,2008,14(7):582-590. doi: 10.1111/j.1601-0825.2007.01424.x [20] TRONCHIN G, PIHET M, LOPES-BEZERRAL M, et al. Adherence mechanisms in human pathogenic fungi[J]. Med Mycol,2008,46(8):749-772. doi: 10.1080/13693780802206435 [21] NOBILE C J, FOX E P, NETT J E, et al. A recently evolved transcriptional network controls biofilm development in <italic>Candidaalbicans</italic>[J]. Cell,2012,148(1-2):126-138. doi: 10.1016/j.cell.2011.10.048 -



 下载:
下载: