-
药用植物中的次生代谢物是中药药效物质的主要来源,已知的植物次生代谢物生物合成途径有乙酸-丙二酸途径、异戊二烯途径、莽草酸途径等[1],探究植物生物合成的调控因素不仅能提升药材的品质,也为中药有效成分体外合成的工业化提供可能。
植物昼夜节律钟是植物体内应对光照、温度等外界因素随昼夜节律性改变而进化出的一套适应机制[2],对植物生长发育具有不可或缺的作用。大量研究表明,如黄酮类化合物合成的相关结构基因表达,也具有明显的昼夜节律性特点,受昼夜节律钟调控[3-4]。昼夜节律钟的核心部分中央振荡器是MYB蛋白LHY(late elongated hypocotyl)、CCA1(circadian clock associated 1)和伪应答调控蛋白家族(PRRs,pseudo-response regulators)组成,对维持植物昼夜节律的稳定至关重要[5-6]。
PRRs基因都带有2个保守的结构域,氨基端的响应接受结构域(receiver-like domain,RLD),其结构上与磷酸接受域相似,羧基端带有的CCT(Constans/Constans-like/TOC1)结构域,这2个结构域被一个保守程度不高的“可变域”所分隔[7-9]。目前研究发现在CO、CO-like以及TOC1基因中也有带此类结构域,对于植物开花进程有着重要作用[10-11]。
研究表明PRRs家族基因具有增加植物抗逆性,影响植物生物量的积累[12-14],以及调控花发育及衰老等作用[15]。目前对模式植物中PRRs基因的研究较多,如在拟南芥以及水稻中的PRRs基因证明具有调控开花周期的作用[17],但药用植物中PRRs基因的研究则罕见报道。
中药红花(Carthami flos)是菊科植物红花(Carthamus tinctorius L.)的干燥花,有活血化瘀的功效。研究表明,红花主要药效物质为黄酮类化合物,如羟基红花黄色素A(HSYA)、红花素、槲皮素、山奈酚、野黄芩苷[18]等,目前已有对红花黄酮类化合物生物合成的关键基因查尔酮合酶、查尔酮异构酶、糖基转移酶等多种研究[19-20],但调控红花中黄酮类化合物生物合成途径的基因未完全明确。验证昼夜节律钟调控红花中黄酮类化合物的生物合成对提升红花品质意义重大。
本研究依据前期红花转录组数据库基因注释筛选昼夜节律相关基因,并与红花黄酮类化合物的积累量进行相关分析,得到具有调控红花黄酮类化合物生物合成功能的昼夜节律基因,通过qPCR、液质联用(UHPLC-MS)等方法以期阐释PRRs基因的特征与功能,为进一步研究昼夜节律钟调控红花黄酮类化合物的生物合成积累资料。
-
植物材料:云南巍山红花品系(编号ZHH0119),种植于海军军医大学药学院温室,温室条件:恒温25 ℃,昼夜节律为16 h光照/8 h黑暗。仪器与试剂:荧光qPCR仪:ABI7500;Phanta Max Master Mix高保真酶,Trans Top green qPCR super mix,Trans one-step cDNA synthesis super mix逆转录试剂盒(北京全式金公司);Meitler Toledo电子天平(十万分之一量程);高效液相色谱仪:Agilent1290 Infinty LC system; 质谱仪:Agilent 6538 Accurate Mass。
-
取用新鲜红花的花冠,根、茎、叶约100 mg研磨成粉。依据Plant Zol说明书提取总RNA,并用紫外分光光度计测定总RNA浓度与质量,样品的A260/A280在1.9~2.1之内可认为符合后续实验要求。将其作为模板逆转录合成cDNA第一链,−20 ℃保存备用。
-
基于红花转录组数据库,结合基因注释筛选出昼夜节律相关基因,通过红花花冠表达谱获取表达量,与红花代谢组数据库中不同花期黄酮类化合物的积累量进行Pearson分析,获取与黄酮类化合物积累量相关系数r≥0.7的昼夜节律基因进行生物信息学分析,设计引物(表1)进行克隆。PCR产物进行凝胶电泳,回收含有目的条带的凝胶,连接载体后转化大肠杆菌感受态细胞,LBA平板培养,挑取阳性克隆送至上海生工生物工程有限公司测序。
表 1 全长克隆引物
引物名称 引物序列 PRR1-F ACCTCAAGGGCCACTGGTTC PRR1-R GTAACAAAAGACTTTCTGAA -
在NCBI网站用BLAST在线分析红花PRR1全长序列以及编码蛋白的氨基酸序列进行比对以及同源性分析;在ExPASy在线工具SOMPA得出所编码蛋白的二级结构特征;通过Swiss-Model同源建模预测蛋白三级结构;在ClustalX 2.1软件对其编码蛋白的氨基酸序列与同源蛋白的氨基酸序列进行多重比对分析;使用相邻节点算法(Neighbor-Joining)构建系统进化树,自展分析法(Bootstrap analysis)进行1 000次重复验证进化树可靠性。
-
以红花根、茎、叶、花4个部位;花冠开花前3 d(Ⅰ期)、开花1 d(Ⅱ期)、开花3 d(Ⅲ期)、开花5 d(Ⅳ期)4个时期[12]以及1 d中8个时间点(6:00、9:00、12:00、15:00、18:00、21:00、0:00、3:00)的花冠第一链cDNA为模板,设计RT-qPCR引物,以Ct60s(KJ634810)作为内参基因,进行qPCR实验,每个样品设3个复孔。
-
取红花Ⅲ期单日内4个时间点(9:00、12:00、15:00、21:00)的花冠烘干至恒重,精密称取5.00 mg置1.5 ml离心管,再加入精密量取的1 ml 60%甲醇,室温放置12 h后超声处理40 min,12 000 r/min,10 min离心取上清液,UHPLC-MS在正、负离子模式下进行检测(表2),参比离子:正离子模式为121.050 9,922.009 8;负离子模式为119.036 3,966.000 7,数据采集与分析使用Agilent MassHunter Analysis4.0软件。以柚皮素、芹菜素、槲皮素、HSYA、山奈酚、红花素、山奈酚-3-O-葡萄糖苷以及野黄芩素为对照品。
表 2 UHPLC-MS分析方法
色谱条件 质谱条件 色谱柱:Agilent ZORBAX C18(3.5 μm,2.1 mm ×100 mm) 毛细管电压:3.5 kV 流动相:A:0.1%甲酸-水,B:乙腈 锥孔电压:60 V 流速:0.4 ml/min 离子源温度:350 ℃ 柱温:40 ℃ 碎裂电压:120 V 进样量:0.4 ml 采集范围:50~1 100 m/z 梯度洗脱:0 min,5%B; 4 min,20%B;
6 min, 21% B; 9 min, 26% B; 11 min, 40% B; 15 min, 80% B; 17 min, 95% B -
使用STRING数据库对CtPRR1基因进行蛋白互作网络分析,可信区间设置为0.4。利用Python下的networkX将互作基因进行可视化。
-
将红花cDNA均一化处理得到红花酵母双杂cDNA文库,与载体PGADT7进行同源克隆,产物转化大肠杆菌感受态细胞,涂布LBA平板,37 ℃培养过夜,挑取单克隆菌落进行菌液PCR验证
-
根据PRR1 ORF设计同源引物引入BamHI、XhoI酶切位点。PCR产物经电泳后回收,连接酶切载体pGBKT7转入大肠杆菌,测序验证后完成pGBKT7-PRR1 bait载体的构建。
将pGBKT7-PRR1bait质粒通过PEG/LiAc法转化酵母感受态细胞,涂布SD平板,28 ℃培养,挑取单克隆进行菌液PCR鉴定。将阳性单克隆酵母菌液划线涂布含有X-α-gal和Aba的平板。
-
将酵母感受态细胞、载体DNA、红花酵母双杂cDNA文库质粒按共转法混合处理后均匀涂布于SD/-His/-Trp/-Ura三缺平板培养,挑取单克隆转移至含有X-α-gal的SD/-His/-Trp/-Ura培养基,扩增阳性质粒用于测序以及酵母双杂验证。
-
实验结果经SPSS19.0软件处理,计量资料统一表示为(
${{\bar x}} \pm {{s}}$ ),组间比较采用ANOVA分析,以P<0.05为差异具有统计学意义。 -
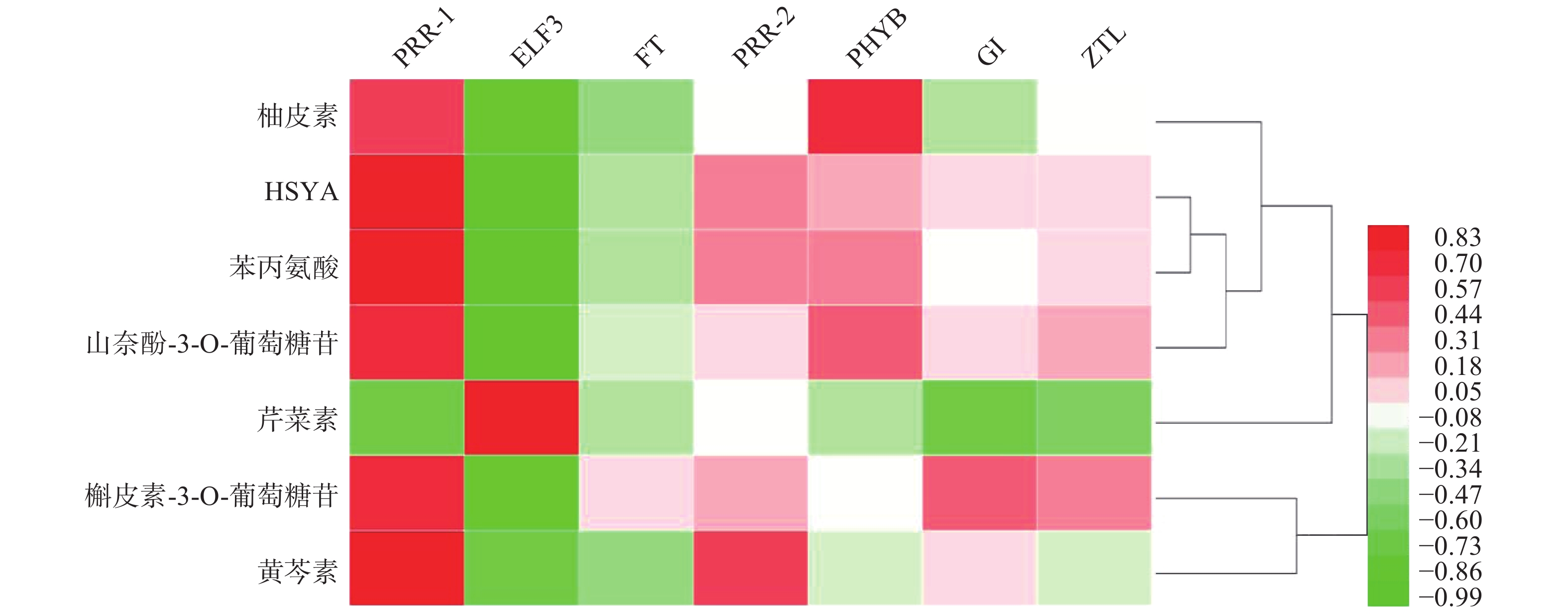
基于红花花冠转录组数据库的基因注释,检索出PRR1、PRR2、ELF3、FT、PHYB、GI、ZTL7个昼夜节律基因,将其在花冠不同时期的表达量与红花代谢组数据库中柚皮素、HYSA、苯丙氨酸、山奈酚-3-O-葡萄糖苷、芹菜素、槲皮苷、野黄芩素、槲皮素-3-葡萄糖苷7个化合物含量进行PEARSON相关性分析,如图1所示,PRR1与红花中主要黄酮类成分的积累量具有良好的相关性(r≥0.7)。
-
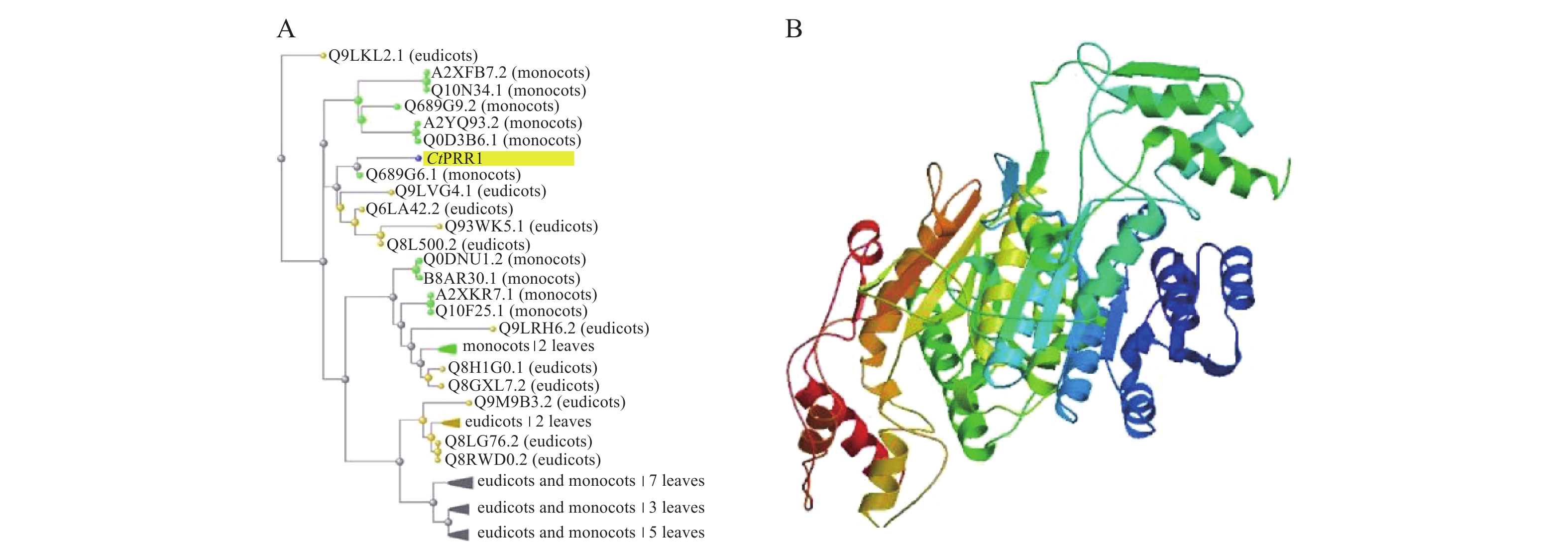
在红花全长转录组数据库中得到的PRR1基因全长序列3 201 bp,ORF FINDER结果显示开放阅读框1 549~2 814 bp,编码421个氨基酸,命名为CtPRR1(GenBank登录号:MW492035),将全长序列进行BLAST,系统发育进化树分析如图2A,CtPRR1氨基酸序列与水稻(Oryza sativa L.)OsPRR73氨基酸序列(A2XFB7.2)同源性最高。Prot-param分析PRR1基因所编码的蛋白质分子式C1900H3039N611O653S15,分子量为45 300,理论pI=8.52,对PRR蛋白质三维结构预测如图2B,Prot Scale分析表明预测PRR1蛋白为亲水性蛋白,无信号肽属非分泌蛋白;蛋白跨膜性分析预测PRR不含有跨膜区域,为非跨膜蛋白。
-
qPCR结果表明(图3A)在红花不同部位中,CtPRR1基因在花中的表达量最高,且与根、茎、叶都有显著性差异(P<0.05),对红花花冠Ⅰ期、Ⅱ期、Ⅲ期、Ⅳ期的CtPRR1基因的表达情况进行分析,CtPRR1在Ⅰ期花冠中表达含量最低;Ⅱ期表达水平略有上升,与Ⅰ期无显著性差异;Ⅲ期花冠中表达量明显上升,相比Ⅰ,Ⅱ期具有显著性差异,Ⅳ期花冠中的CtPRR1基因表达水平略微下降(图3B),表明CtPRR1基因在Ⅲ期花冠中转录水平最高,CtPRR1在不同时间表达量的变化与红花黄酮类化合物积累量的变化相符[22]。对红花8个时间点的花冠CtPRR1表达量分析发现CtPRR1表达量在日间6:00至18:00逐渐升高,18:00达峰值,晚间21:00至2:00逐渐下降,3:00最低,表达水平在白天与夜晚两个连续的时间周期内呈现昼夜节律性(图3C)。
-
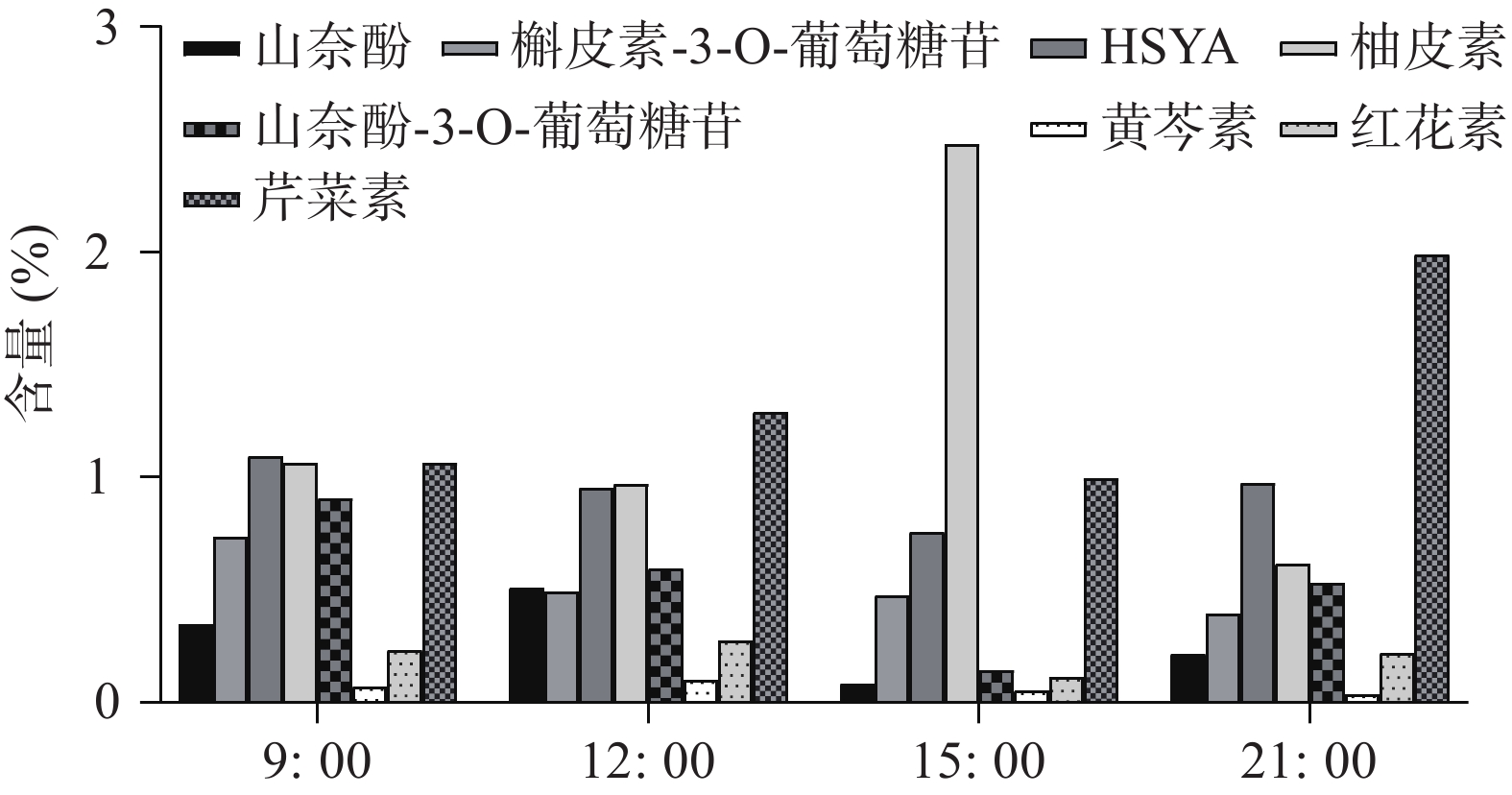
UHPLC-MS检测黄酮类化合物在单日内不同时间的含量变化(图4),除柚皮素外的7个化合物在白天含量逐渐降低,而在晚间含量又升至较高的水平,柚皮素则相反在白天升高,晚间下降,但8个化合物含量变化也呈现昼夜节律性,与同时间点CtPRR1的表达量进行PEARSON分析得到红花素(r=−0.9485)、山奈酚(r=−0.9423)、野黄芩苷(r=−0.9504)、HSYA(r=−0.8372)、山奈酚-3-O-葡萄糖苷(r=−0.8792)、柚皮素(r=0.7415)、芹菜素(r=−0.6652)、槲皮素(r=−0.4876),目前研究认为柚皮素在红花黄酮类化合物生物合成途径的上游[20],而CtPRR1与柚皮素呈正相关,与其他化合物呈负相关,说明CtPRR1对整体黄酮类化合物生物合成具有负调控作用而导致柚皮素的积累量增多,进一步说明CtPRR1参与调控红花黄酮类化合物的合成。
-
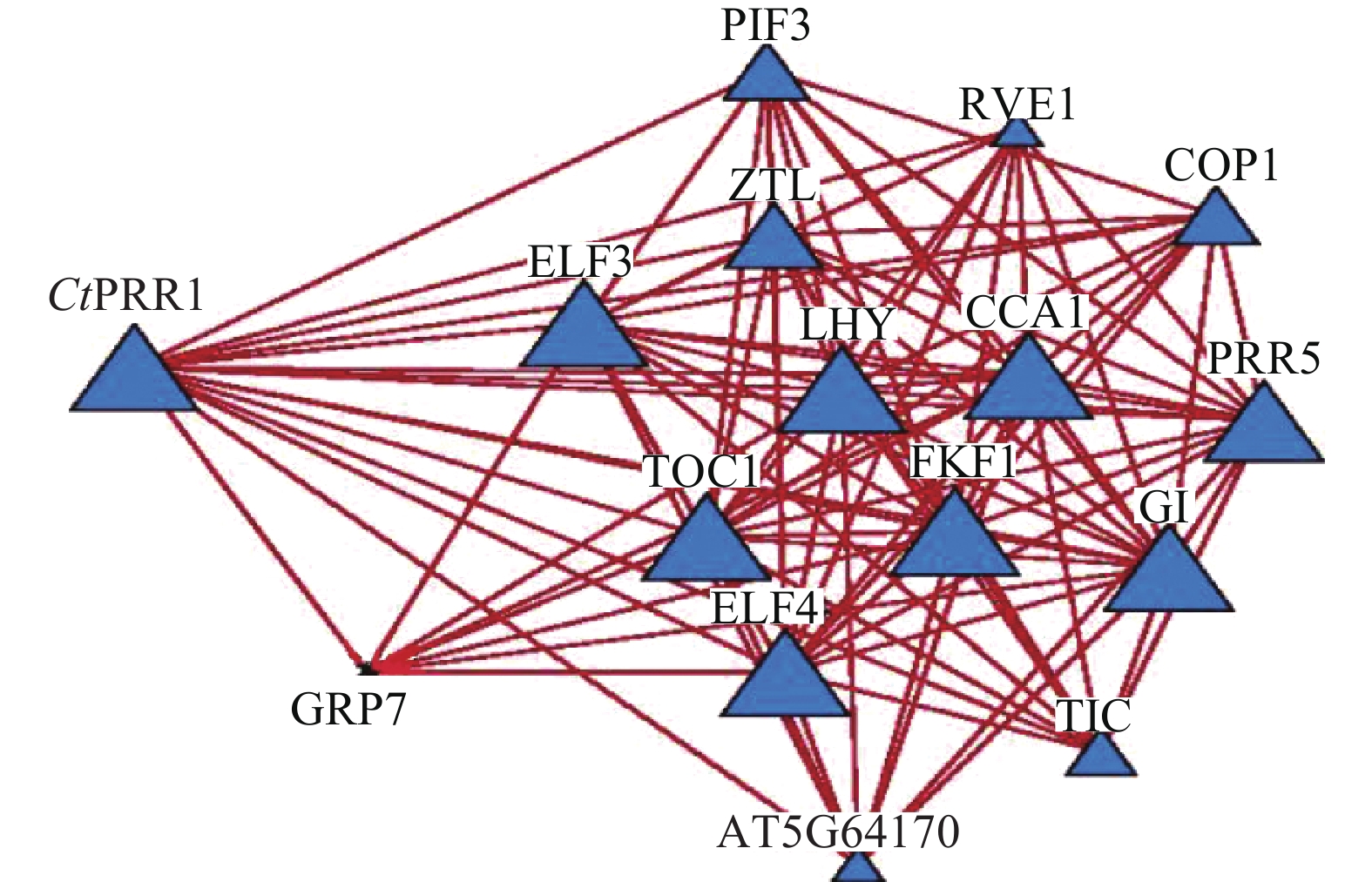
STRING数据库预测红花转录组数据库中CtPRR1的互作蛋白,获得互作蛋白TOC1、PIF3、COP1、ZTL、LHY、ELF4、ELF3、CCA1、GI、PRR5、GRP7、TIC、FKF1、RVE114个,互作关系网络如图5。与PRRs基因共同参与调控植物生长发育,调控植物次生代谢途径。由此获得了较为完善的红花昼夜节律核心元件系统,为进一步探索昼夜节律调控红花次生代谢的分子机制提供了依据。
通过酵母双杂实验发现,转入pGBKT7-PRR bait质粒的酵母细胞在28 ℃生长4 d后情况正常。带有pGBKT7-PRR bait载体的酵母细胞与构建的红花cDNA文库质粒共转后,28 ℃培养2 d后可以观察到蓝色菌斑出现,4 d后出现直径约2 mm的蓝色单克隆菌斑(图6),菌液PCR鉴定为阳性克隆。结果如表3所示,有2个热休克蛋白,3个AP2转录因子,热休克蛋白具有增强植物抗逆性的功能,AP2转录因子广泛参与植物生长发育,调控体内次生代谢。
表 3 酵母双杂筛选互作蛋白
序号 蛋白家族名称 预测功能 1 热休克蛋白 广泛参与植物响应外界环境胁迫,生物合成以及生长发育相关的转录调控 2 热休克蛋白 3 AP2转录因子 4 AP2转录因子 5 AP2转录因子 -
红花作为常用的活血化瘀中药,对其主要药效物质黄酮类化合物的生物合成途径及调控机制的研究越来越多[20],但目前红花黄酮类化合物生物合成途径及调控机制仍未阐明。
本研究首次克隆了一个PRRs家族基因CtPRR1,生物信息学分析表明其与水稻、拟南芥等其他物种中的PRRs家族基因序列高度相关,说明红花中的PRRs基因具有高度保守性。CtPRR1基因主要在花中表达且开花后第3天时表达量最高,与红花不同花期黄酮类化合物的累积规律一致,存在显著相关。我们认为,CtPRR1调控了红花黄酮类化合物的生物合成。进一步的研究发现,CtPRR1的单日表达量在日间逐渐升高,晚间逐渐下降;随着CtPRR1在单日表达量的升高,芹菜素、槲皮素、HSYA、山奈酚、红花素、山奈酚-3-O-葡萄糖苷以及野黄芩素积累量为白天逐渐降低,晚间逐渐升高。CtPRR1对红花这些黄酮类成分的昼夜节律性积累积起负调节作用;唯CtPRR1与柚皮素的积累量呈正相关,可能与柚皮素处于黄酮生物合成途径的较上游以及参与其他代谢过程并受到其他调控基因的影响有关。
PRRs与CCA1、LHY基因作为昼夜节律系统中的核心元件,对多种植物的昼夜节律钟输出途径中与黄酮合成结构酶基因具有相互作用[22-23],本研究发现红花CtPRR1可能受2个热休克蛋白,3个AP2转录因子的影响对黄酮化合物的积累起负调节作用,丰富了昼夜节律基因调控黄酮类化合物机制的研究资料。
本研究结果为深入研究红花昼夜节律基因对黄酮生物合成途径的调控机制提供了资料。下一步的工作,我们将采用基因过表达以及基因敲除技术结合代谢物分析,进一步验证CtPRR1调控红花黄酮类化合物生物合成的功能及调控网络,为阐明CtPRR1的功能提供重要依据。
Characterization and function study of circadian rhythm gene CtPRR1 in Carthamus tinctorius L.
-
摘要:
目的 探究昼夜节律基因对红花黄酮类物质生物合成的影响及机制。 方法 基于红花花冠转录组及代谢组数据库筛选潜在调控红花黄酮类化合物生物合成的昼夜节律基因;用qPCR测定红花各部位以及花冠单日不同时间点昼夜节律基因的表达量,液质联用测定黄酮类化合物的积累量,并分析二者的相关性;酵母双杂交实验验证昼夜节律基因的互作蛋白。 结果 获得7个昼夜节律基因PRR1、PRR2、ELF3、FT、PHYB、GI、ZTL,其中PRR1基因与黄酮类化合物积累量呈正相关(r≥0.7)。PRR1全长3 201 bp,编码421个氨基酸,与水稻OsPRR73基因高度同源,将其命名为CtPRR1(GenBank登录号:MW492035)。CtPRR1主要在花中表达,表达量在日间逐渐升高,晚间逐渐下降;黄酮类化合物芹菜素、槲皮素、HSYA、山奈酚、Carthamin、山奈酚-3-O-葡萄糖苷以及野黄芩素的含量为白天逐渐降低,晚间逐渐升高,二者都有昼夜节律性且呈负相关(r≥−0.7)。酵母双杂实验得到2个热休克蛋白、3个AP2转录因子。 结论 CtPRR1对红花黄酮类成分的昼夜节律性积累起负调节作用;CtPRR1可能受这些互作蛋白的影响调控红花黄酮类成分的昼夜节律性积累。 Abstract:Objective To explore the effect of circadian rhythm genes on flavonoids biosynthesis in safflower and its molecular mechanism. Methods Based on the transcriptome and metabolomic database of safflower corolla, we screened the circadian rhythm genes that correlate with biosynthesis of flavonoids in safflower. qPCR was used to quantify the expressions of circadian rhythm genes in different flowering stages at different time points in a single day. LC-MS was performed to determine the accumulation of flavonoids. The correlation between them was analyzed as well. Yeast Two-Hybrid experiment was used to verify the interactive proteins of these genes. Results Seven circadian rhythm genes PRR1, PRR2, ELF3, FT, PHYB, GI and ZTL were obtained. PRR1 gene was positively correlated with flavonoids accumulation (r≥0.7). The full length of PRR1 is 3 201 bp, encoding 421 amino acids, which is highly homologous with rice OsPRR73 gene and named as CtPRR1 (GenBank accession number: MW492035). CtPRR1 was mainly expressed in flowers, and the expression level increased in the daytime and declined in the evening gradually. Correspondingly, the content of flavonoids showed an opposite variation. Both of them displayed a circadian rhythm with a negative correlation (r≥−0.7). In addition, 2 heat shock proteins along with 3 AP2 transcription factors interacting with CtPRR1 protein were obtained via Yeast Two-Hybrid experiment. Conclusion CtPRR1 negatively regulated the safflower flavonoids accumulation in a circadian rhythm way, which may be affected by these interacting proteins. -
Key words:
- Carthami flos /
- circadian gene /
- flavonoids /
- interaction protein
-
补体系统是人体重要的免疫防御系统之一,是由30多种广泛存在于血清、组织液和细胞膜表面的蛋白质组成的,具有精密调控机制的蛋白质反应系统,其主要通过3种途径激活:经典途径、旁路途径和甘露糖结合凝集素途径。补体系统正常激活,可在靶细胞上形成膜攻击复合物,导致靶细胞的溶解,补体的这一功能在机体的免疫系统中起重要的防御和免疫监视作用,对抵御外来微生物的入侵和维持机体平衡有重要的作用。然而该系统的过度激活将释放炎性过敏毒素C3a和C5a,具有化学诱导作用的C5a能趋化嗜中性粒细胞、中核细胞和嗜酸性粒细胞,这些细胞释放蛋白酶和具有趋化作用细胞因子,进一步聚集T、B淋巴细胞和其他炎性细胞,从而促进炎症反应的发生,引起系统性红斑狼疮、类风湿性关节炎、动脉粥样硬化、肾小球肾炎等[1-2]。近年来已有研究表明[3],补体系统的激活是类风湿性关节炎中慢性滑膜炎的发病因素之一。因此,抑制补体系统的过度激活可能是治疗类风湿性关节炎的重要机制之一。
三色片为复旦大学附属中山医院的院内制剂,由雷公藤、黄芪和丹参三味药材按1∶1∶1的比例配伍组成,在临床上用于治疗类风湿性关节炎、系统性红斑狼疮、银屑病和湿疹等结缔组织疾病。我院临床医生在长期的医疗实践中总结出来的经验方,效果显著[4]。组方中雷公藤,性味辛寒,有大毒,归肝、肾经,具有清热解毒、活血化瘀、通络止痛、杀虫止痒等功效。现代研究表明,雷公藤内酯醇对大鼠脑皮质内注射β-淀粉酶后补体C1q和C3的表达有抑制作用,表明雷公藤对补体系统有抑制作用,目前临床上广泛用于治疗类风湿性关节炎、系统性红斑狼疮、银屑病和湿疹等结缔组织疾病[5]。组方中的黄芪用于脾肺气血或中气下陷之症、卫气虚所致表虚自汗、气虚血滞导致的肢体麻木、关节痹痛等症,可联合治疗类风湿性关节炎[6]。黄芪在治疗2型糖尿病大鼠的研究中发现其能降低补体C3的水平,表明其对补体系统具有一定的调节作用[7-8]。丹参是最常用的活血化瘀中药之一,具有祛瘀止痛,养血安神的功效,现代药理学研究表明其还具有保护肝脏的功能[9],可拮抗雷公藤的肝毒性。本研究通过经典途径抗补体活性测定方法筛选出三色片醇提物的乙酸乙酯部位抗补体活性最佳,并采用UPLC-Q-TOF-MS法对该部位的化学成分进行结构表征,为三色片抗补体活性药效物质基础及治疗补体过度激活相关疾病提供科学依据。
1. 仪器、试剂与材料
Tripie TOF5600+型四级杆-飞行时间串联质谱仪,配备电喷雾电离源和CDS自动校正系统(美国Applied Biosystems公司);Peak view2.2和Master view1.1数据处理系统(美国Applied Biosystems公司);LC-30A超高效液相色谱仪,包括高压输液泵,自动进样器,柱温箱和在线脱气机(日本岛津公司);KQ5200E型超声清洗器(昆山市超声仪器有限公司);甲醇、乙腈(色谱纯,德国Merck公司);甲酸(色谱纯,美国Sigma-Aldrich公司); 蒸馏水(娃哈哈集团);三色片提取物由作者自制,现样品存放于复旦大学附属中山医院药剂科(SSP2018);补体、溶血素(自制);毛蕊异黄酮(批号:ST088101),雷公藤甲素(批号:ST020501),雷公藤内酯酮(批号:ST049901),丹参酮II A(ST014601)、黄芪甲苷(ST001601)(纯度≥ 98%,均购自上海斯丹德生物技术有限公司)。
2. 方法
2.1 三色片醇提物及各极性部位的制备
雷公藤、黄芪和丹参三味药材按1∶1∶1配伍,其中,黄芪和丹参加6倍量的水浸泡2 h后,煎煮2次,第一次1.5 h,第二次加水4倍量煎煮1 h,煎液滤过,合并滤液并浓缩至相对密度为1.10~1.20(70 ℃),加入2倍量的乙醇,静置沉淀24 h,取上清液备用。雷公藤分别加4倍量的乙醇加热回流2次,每次1.5 h,合并提取液,滤过,加入上述备用药液,混匀,回收乙醇至无醇味,浓缩后即得三色片醇提物,经现有的质量标准检验为制备三色片制剂合格的提取物。精密称取三色片醇提物2.0 g,置于100 ml萃取瓶中,加25 ml蒸馏水溶解后,用等量的石油醚、乙酸乙酯和正丁醇进行萃取,浓缩干燥后,放冷至室温,得到三色片醇提物的石油醚部位0.36 g,乙酸乙酯部位0.42 g,正丁醇部位0.56 g和水溶性部位。
2.2 经典途径的抗补体活性测定
取各极性部位样品2 mg溶于DMSO,采用BBS缓冲液稀释成不同浓度的样品,并加入临界浓度的补体(1∶80稀释的豚鼠血清),溶血素和2%绵羊红细胞(SRBC)。37 ℃水浴30 min,离心后取上清液在405 nm波长下测定吸光度(A)值。同时设置中药对照组(将等量的中药提取物加入BBS缓冲液中,用于测定中药本底A值)、补体组(取临界浓度的补体直接加入适量的BBS缓冲液、溶血素和2%SRBC,用于测定临界浓度补体所造成红细胞溶血的A值)和全溶血组(将2%SRBC加入水中使之全溶血,用于观察补体组是否达到或接近全溶血水平),并以肝素作为阳性对照组,计算溶血抑制率。以供试品浓度为横坐标(X),溶血抑制率为纵坐标(Y),计算CH50(经典途径50%抑制溶血所需供试品浓度)。溶血抑制率=1−(A中药−A中药对照)/A全溶血。
2.3 不同浓度的样品色谱与质谱条件
2.3.1 色谱条件
色谱柱为ACQUITY UPLC BEH C18(2.1 mm×100 mm,1.7 μm);流动相0.1%甲酸和水溶液(A)−乙腈(B);梯度洗脱:0~9 min,10%~23% B;9~13 min,23% B;13~28 min,23%~40% B;28~32 min,40%~50% B;32~37 min,50%~100% B;37~42 min,100% B;42~42.1 min,10%B;42.1~50 min,10% B;流速为0.25 ml/min,柱温为35 ℃;进样量为2 μl。
2.3.2 质谱条件
在正/负离子模式,离子源选择电喷雾离子化源(ESI);使用m/z 50~1250扫描范围;碰撞能量35 eV,碰撞能量叠加(35±15)eV;喷雾电压5 500 V;雾化气温度550 ℃;去簇电压100 V;雾化气和辅助气均为50 psi;气帘气25 psi;数据采集时间50 min;采用母离子触发的子离子(TOF-MS-IDA-MS/MS)扫描方式;多重质量亏损和动态背景扣除为触发二级的条件,满足该条件进行二级扫描。
2.4 对照品溶液的制备
精密称取毛蕊异黄酮、雷公藤甲素、雷公藤内酯酮、丹参酮Ⅱ A和黄芪甲苷对照品1.0 mg,加甲醇2 ml,溶解,摇匀,即得各对照品溶液。
2.5 供试品溶液的制备
取三色片醇提物的乙酸乙酯部位样品0.2 g,置于10 ml量瓶中,加入70%甲醇5 ml,超声处理(功率250 W,频率40 kHz)30 min,放冷至室温,70%甲醇定容至刻度,摇匀,滤过,取续滤液,即得供试品溶液。
2.6 三色片中化学成分数据库的建立
根据三色片中各药材化学成分研究文献,收集3种药材所含化合物成分的基本信息,包括化合物名称、分子式、精确分子量、准分子离子峰和碎片离子峰。通过精确分子量匹配,对照品的保留时间,二级谱所得到的离子碎片与文献报道进行比对,最终确定化合物的结构。
3. 结果与分析
3.1 三色片醇提物各极性部位的抗补体活性
分别对三色片醇提物的石油醚部位、乙酸乙酯部位和正丁醇部位进行经典途径的抗补体活性测定,以肝素为对照品,结果发现乙酸乙酯部位的抗补体活性最好,其抗补体活性略低于肝素钠,其次是正丁醇部位,结果见表1。
表 1 三色片提取物不同部位抗补体活性测定编号 研究对象 抗补体活性(CH50,μg/ml) 1 肝素 14.4±1.2 2 三色片-石油醚部位 − 3 三色片-乙酸乙酯部位 233.9±10.1 4 三色片-正丁醇部位 344.0±14.5 注:“—”表示该部位无抗补体活性。 3.2 三色片醇提物乙酸乙酯部位的UPLC-Q-TOF-MS分析
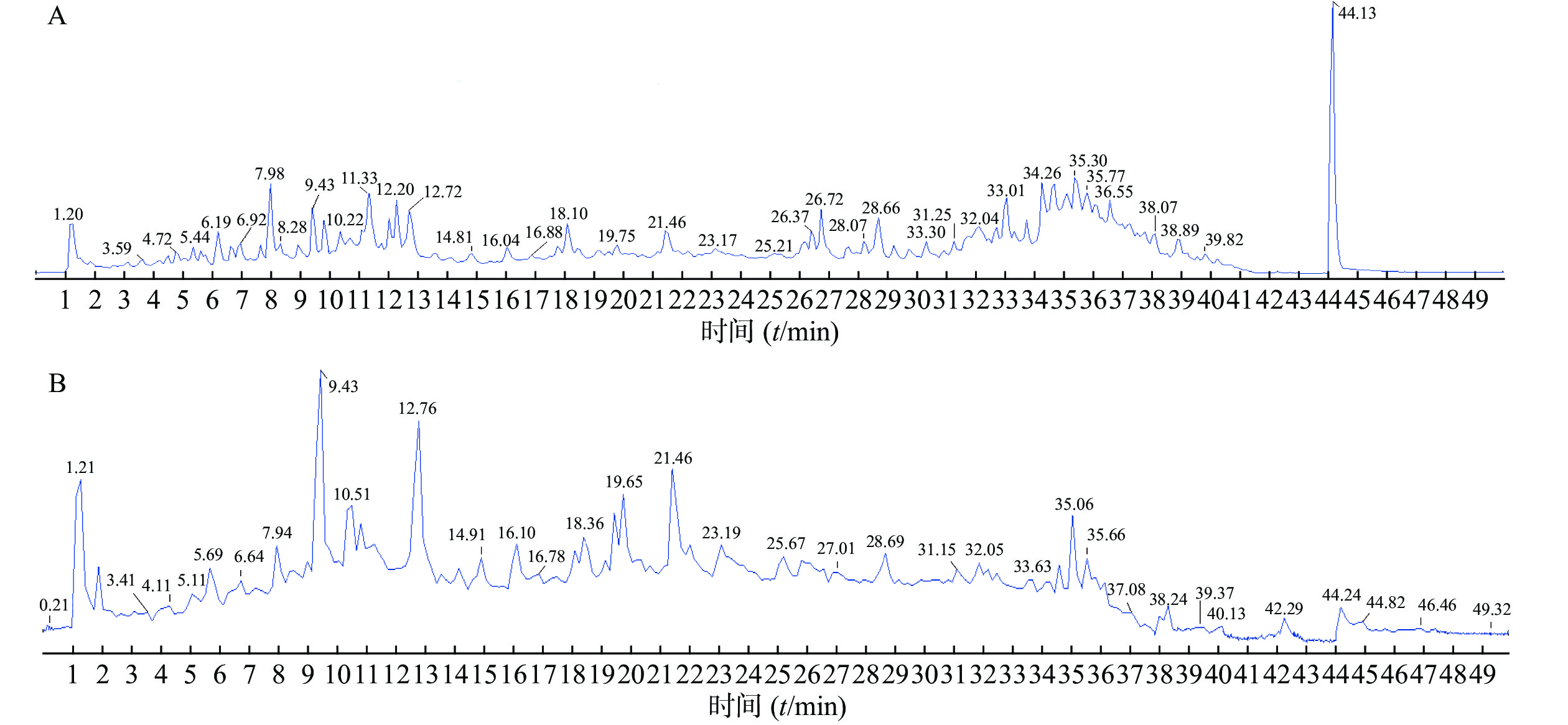
精密吸取对照品溶液和供试品溶液2 μl,采用“2.1”项下的色谱与质谱条件对样品进行分析,通过正、负离子全扫描,获得正、负离子模式下的总离子流图,见图1。
通过与对照品比对,分子离子峰质谱数据解析,与参考文献比对,共鉴定出三色片醇提物乙酸乙酯部位42个化合物,结果见表2。
表 2 三色片提取物中各成分主要碎片离子及谱峰归属化合物
编号tR/min 分子式 理论值(m/z) 模式 实测值(m/z) 误差(×10−6) 碎片离子(m/z) 化合物名称 参考文献 1 3.54 C7H6O3 139.039 0 [M+H]+ 139.039 4 3.0 121.028 7 原儿茶醛 [10] 2 4.76 C21H27N3O3 370.212 5 [M+H]+ 370.214 3 4.8 249.124 6,160.112 6,95.013 3,166.086 6,100.076 2,91.054 8 南蛇藤糠酰胺碱 [11] 3 6.63 C22H22O10 447.128 6 [M+H]+ 447.130 4 4.2 285.077 5,270.053 5,253.050 8,225.055 6,137.023 5 毛蕊异黄酮-7-O-β-D-葡萄糖苷 [12] 4 7.04 C23H29N3O2 380.233 3 [M+H]+ 380.235 1 4.8 176.106 9,160.112 6,105.033 8,100.076 5 苯代南蛇碱 [11] 5 9.41 C9H10O5 197.045 6 [M-H]− 197.044 7 −4.2 179.038 3,135.044 3 丹参素 [10] 6 9.42 C9H8O4 179.035 0 [M-H]− 179.034 2 4.7 135.044 8 咖啡酸 [13] 7 11.16 C16H12O4 431.133 7 [M+H]+ 431.136 3 6.1 269.082 6,253.050 3,225.055 5,213.091 7,197.060 2,136.014 6,118.041 7 芒柄花苷 [12] 8 12.71 C16H12O5 285.075 8 [M+H]+ 285.077 4 5.7 270.053 4,253.050 3,225.055 3,137.023 5 毛蕊异黄酮* [12] 9 12.76 C17H16O5 301.107 1 [M+H]+ 301.109 0 6.4 167.070 8,152.047 3,147.043 2,105.034 0,123.043 3 astrapterocarpan [12] 10 13.06 C20H24O6 361.164 6 [M+H]+ 361.166 5 5.5 269.154 3,227.108 3,185.096 9,157.101 7,129.070 3,91.054 9 雷公藤甲素* [14-15] 11 13.92 C17H14O6 315.086 3 [M+H]+ 315.088 1 5.6 300.064 7,243.065 5,167.034 2 熊竹素 [16] 12 18.21 C18H12O7 341.065 6 [M+H]+ 341.066 9 3.9 295.060 7,277.050 9,249.056 0 丹酚酸G [17] 13 19.76 C26H20O10 491.098 4 [M-H]− 491.097 0 −2.9 311.054 9,293.044 6,267.064 6,135.044 7 丹酚酸C [18] 14 21.46 C16H12O4 269.080 8 [M+H]+ 269.082 7 4.1 253.015 3,237.052 6,225.055 5,213.092 3,136.015 9,118.041 7,197.060 2 芒柄花素 [12] 15 22.57 C36H45NO17 764.276 0 [M+H]+ 764.278 3 2.9 746.276 1,686.246 3,644.235 1,206.081 7,188.070 9,178.086 5 aquifoliunine E-Ⅲ [14] 16 23.45 C20H22O6 359.148 9 [M+H]+ 359.150 7 4.9 267.138 0,225.019 5,183.079 9,128.061 8,91.054 3 雷公藤内酯酮* [19] 17 24.01 C38H47NO19 822.281 5 [M+H]+ 822.284 1 3.2 804.275 8,204.066 2,176.071 4 alatusinnine [20] 18 25.01 C39H45NO19 832.265 9 [M+H]+ 832.269 0 3.8 804.273 3,194.081 9,176.071 2 hypoglaunine E [11] 19 26.14 C41H68O14 829.458 0 [M+COOH]− 829.460 7 3.3 783.457 9,621.404 3,489.357 2 黄芪甲苷* [14] 20 28.19 C38H47NO18 806.286 6 [M+H]+ 806.290 3 3.8 788.279 5,686.247 0,206.082 1, 178.086 5 雷公藤定宁 E [20] 21 28.65 C39H45NO18 816.271 0 [M+H]+ 816.273 9 3.6 798.261 9,756.250 9,206.081 3,178.086 1,160.075 2 1-去乙酰基雷公藤吉碱 [11] 22 28.70 C43H70O15 871.468 6 [M+COOH]− 871.470 8 2.5 825.470 2,765.448 2,489.356 8 黄芪皂苷Ⅱ [13] 23 29.09 C41H47NO20 874.276 4 [M+H]+ 874.278 5 2.3 856.269 2,846.282 9,828.272 3,674.245 1,204.065 6,176.070 7 雷公藤春碱 [11] 24 29.72 C38H47NO18 806.286 6 [M+H]+ 806.291 2 3.8 788.280 4,686.247 4,206.082 4 peritassine A [20] 25 30.24 C43H70O15 871.468 6 [M+COOH]− 871.470 3 2.5 825.464 1,765.440 5 异黄芪皂苷Ⅱ
(异构体1)[16] 26 30.89 C19H16O4 309.112 1 [M+H]+ 309.114 2 6.7 281.667 0,263.106 0,235.076 7 丹参醛 [21] 27 31.58 C43H70O15 871.468 6 [M+COOH]− 871.470 8 2.5 825.470 2,765.448 0 异黄芪皂苷Ⅱ
(异构体2)[13] 28 32.12 C21H20O4 337.143 4 [M+H]+ 337.142 5 −2.7 309.686 6 丹参新醌丁 [10] 29 32.16 C43H49NO19 884.297 2 [M+H]+ 884.299 7 2.8 856.304 5,674.246 0,204.663 0,176.071 2 雷公藤定碱 [14] 30 32.96 C45H72O16 913.479 1 [M+COOH]− 913.482 4 3.5 867.481 7,825.469 8,807.464 3,765.450 6 黄芪皂苷Ⅰ [16] 31 32.99 C41H47NO19 858.281 5 [M+H]+ 858.285 4 4.6 840.275 7,798.263 8,746.269 1,738.243 5,686.248 0,206.082 5,178.087 1 雷公藤晋碱 [20] 32 33.04 C38H47NO18 806.286 6 [M+H]+ 806.289 7 3.8 788.278 3,686.244 4,206.082 1,728.257 0 卫矛碱 [20] 33 33.70 C45H72O16 913.479 1 [M+COOH]− 913.484 7 3.8 867.478 5,825.283 5,807.458 4,765.432 6 异黄芪皂苷Ⅰ
(异构体1)[16] 34 34.60 C45H72O16 913.479 1 [M+COOH]− 913.483 3 3.8 867.477 8,825.282 1,807.456 4,765.443 2 异黄芪皂苷Ⅰ
(异构体2)[16] 35 34.92 C46H49NO22 968.281 9 [M+H]+ 968.286 3 4.5 856.677 0,838.257 4,684.228 8,204.065 6,178.070 8 雷公藤素B [20] 36 35.01 C43H49NO18 868.302 2 [M+H]+ 868.304 6 2.7 868.364 0,850.295 8,746.268 9,686.247 6,206.082 4,178.087 1 雷公藤次碱 [20] 37 35.02 C41H47NO17 826.291 7 [M+H]+ 826.295 1 4.2 808.285 3,748.264 0,206.082 2,178.086 8 tripterygiumine Ⅰ [20] 38 35.49 C19H20O3 297.148 5 [M+H]+ 297.145 0 4.8 251.144 0,279.139 3,254.054 9,268.110 5,282.126 3 隐丹参酮 [10,17] 39 35.70 C20H28O2 299.201 7 [M-H]− 299.199 6 −6.7 283.168 2,213.090 8,201.916 0, 雷酚萜 [22] 40 35.86 C48H51NO18 930.317 9 [M+H]+ 930.321 3 3.7 912.308 7,310.111 0,206.081 8,188.071 2,178.086 5,105.033 6 ebenifoline E-Ⅱ [20] 41 36.81 C19H18O3 295.132 9 [M+H]+ 295.134 9 4.0 277.124 3,249.127 5,266.095 3,262.097 7,280.109 9 丹参酮Ⅱ A* [10,18,23] 42 37.49 C19H22O2 283.169 3 [M+H]+ 283.169 3 0 265.098 1,240.032 2,223.106 7,195.095 8,181.101 1 丹参新酮 [17,21,24] 注:*表示与对照品鉴定的化合物。 3.2.1 黄酮类化合物结构解析
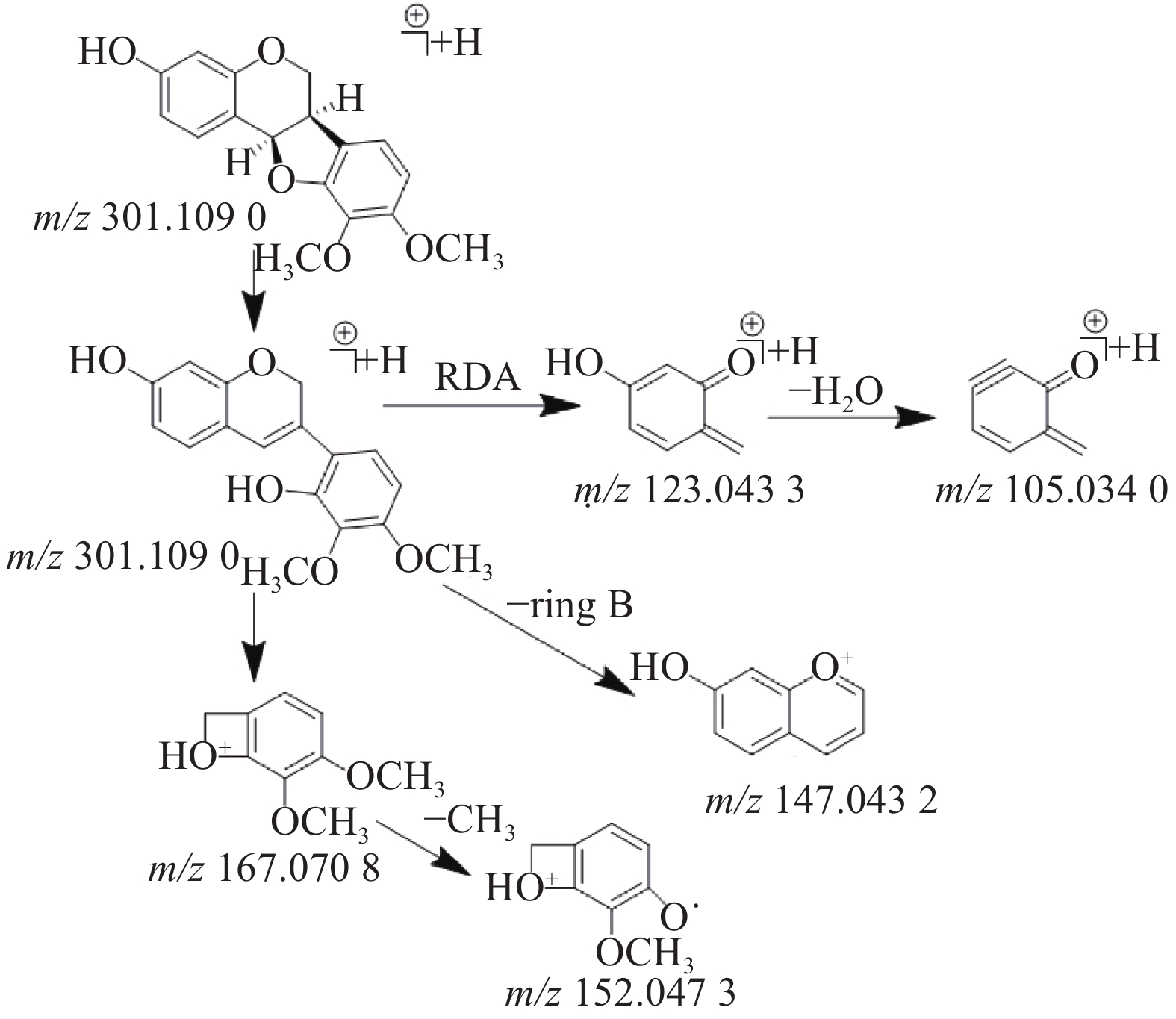
在乙酸乙酯部位中共鉴定出6个黄酮类化合物,其中4个黄酮苷元和2个黄酮苷,苷元为黄酮、异黄酮和紫檀烷,该类化合物在正离子模式下具有较好的响应。二级质谱中黄酮苷元易发生中性丢失,形成[M+H-H2O]+、[M+H-CO]+、[M+H-CH3]+等碎片离子,如在化合物8的二级质谱中可见m/z 270.053 4和m/z 253.050 3,则为m/z 285.077 4分别脱去-CH3和CH3OH形成的[M+H-CH3]+和[M+H-CH3OH]+碎片离子峰,m/z 225.055 3是m/z 253.050 3脱去1分子的CO形成的碎片离子峰,通过对照品的保留时间和参考文献[12]质谱数据比对确定化合物8为毛蕊异黄酮,m/z 137.023 5的碎片离子峰为异黄酮母核C环发生RDA裂解所产生。黄酮苷类易脱去糖基形成较强的分子离子峰,如化合物3(m/z 447.130 4)的二级质谱脱去糖基形成m/z 285.077 5的分子离子峰,并与化合物8(m/z 285.077 4)的二级质谱图非常相似,说明化合物3和化合物8在结构上是相似的,但化合物3的分子量多了162(C6H10O5),通过数据库比对和参考文献[12]推测化合物3则为毛蕊异黄酮-7-O-β-D-葡萄糖苷。化合物9(m/z 301.109 0)通过数据库比对发现两种候选化合物分别为astrapterocarpan和astraisoflavan,二级质谱中主要碎片离子峰为C环裂解产生的含A环和B环片段的碎片离子,其中,m/z 167.070 8为含B环的碎片离子峰且为基峰,进一步脱甲基形成m/z 152.047 3,m/z 123.043 3为含A环的碎片离子峰,进一步脱水形成m/z 105.034 0,m/z 147.043 2为母离子m/z 301.109 0脱去B环形成的碎片离子峰,根据m/z 167.070 8的碎片离子峰为基峰和含有m/z 147.043 2的碎片离子峰这两个特征,结合参考文献[12]的质谱数据,推测该化合物为astrapterocarpan,其相关裂解途径见图2。
3.2.2 三萜皂苷类化合物结构解析
在乙酸乙酯部位中鉴定出7个三萜皂苷类化合物,在负离子模式下均具有较好的响应,一级质谱中产生[M+COOH]−的准分子离子峰,二级质谱中产生较强的[M-H]-碎片离子峰和脱去糖基的较弱的分子离子峰。化合物19在负离子模式下产生的准分子离子峰为[M+COOH]−(m/z 829.458 0),二级质谱中产生m/z 783.457 9[M-H]−峰,脱去1分子六碳糖(C6H10O6)形成m/z 621.404 3的碎片离子峰,m/z 489.357 2则为m/z 621.404 3进一步脱去1分子五碳糖(C5H6O5)后形成的苷元碎片离子峰,推测其苷元为9,19-环阿尔廷烷,通过对照品的保留时间,参考文献[13]的离子碎片比对确定该化合物为黄芪甲苷。
3.2.3 生物碱类化合物结构解析
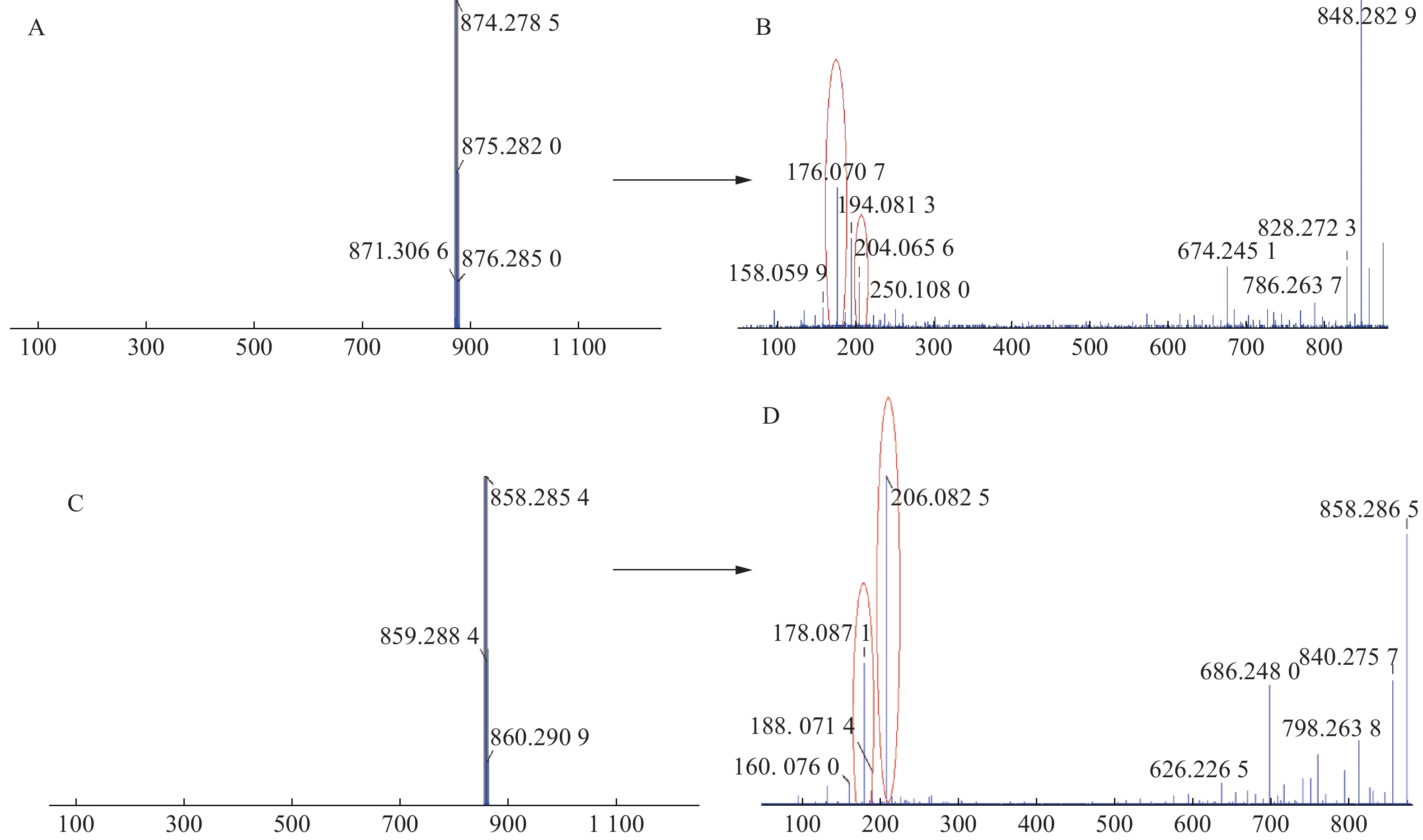
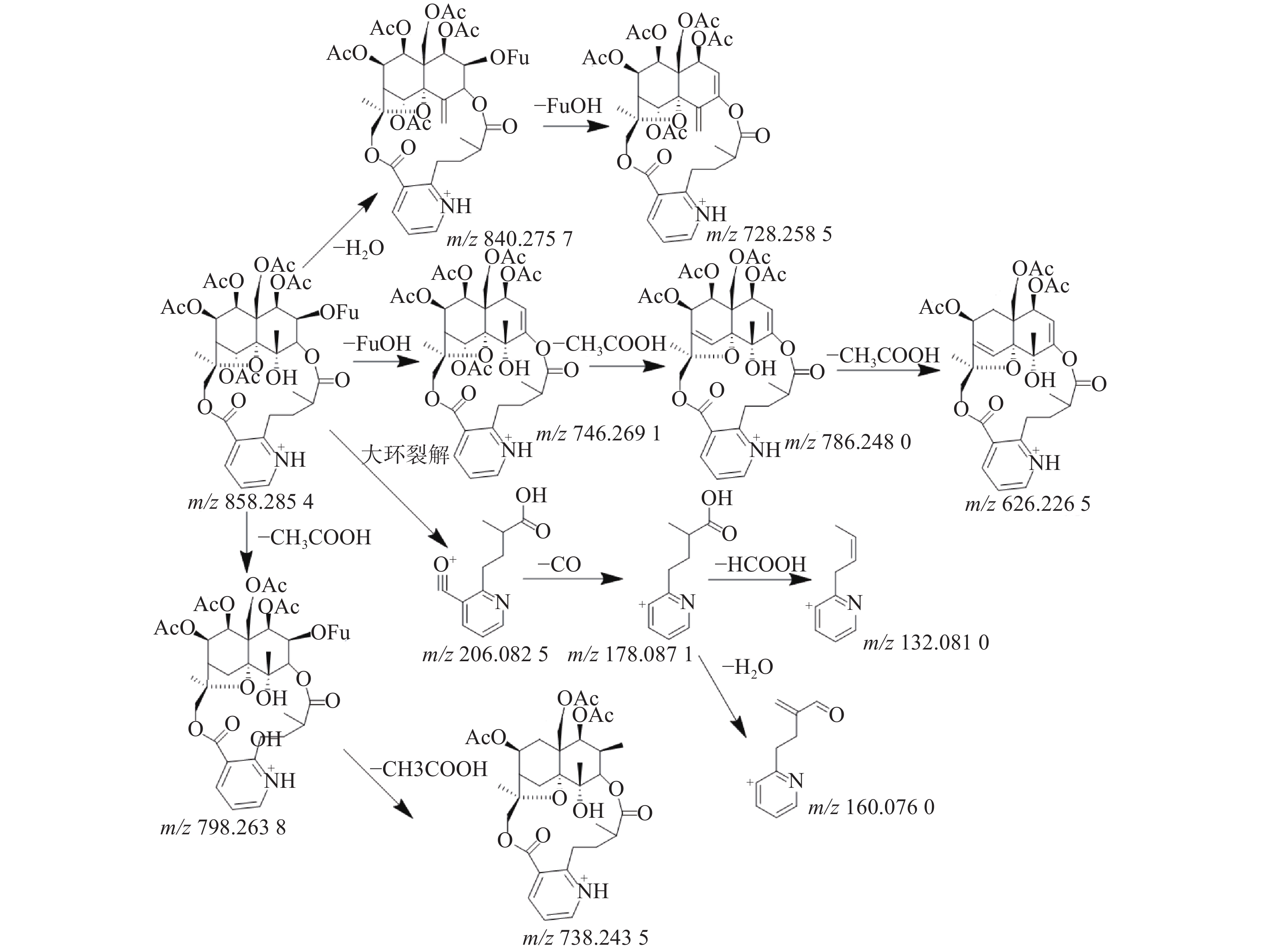
三色片提取物中共鉴定出16个生物碱类化合物,均来自雷公藤药材,在正离子模式下具有较好的响应,一级质谱中产生[M+H]+的准分子离子峰,二级质谱发现该类型的化合物容易脱去H2O、CO和CH3COOH等中性小分子而产生碎片离子峰,多数生物碱含有吡啶二羧酸部位的碎片离子峰。如化合物23在正离子模式下产生m/z 874.278 5的准分子离子峰,二级质谱中产生脱去1分子CO的m/z 846.282 9的基峰,脱去1分子H2O的m/z 856.2692的碎片离子峰和脱去1分子HCOOH的m/z 828.272 3的碎片离子峰,m/z 674.245 1峰为m/z 846.2829脱去C5H4O3侧链和CH3COOH形成的碎片离子峰,m/z 204.065 6峰为大环开裂产生的吡啶二羧酸部分脱水产生的碎片离子,该离子进一步脱羧形成m/z 176.070 7的碎片离子,通过数据库和参考文献[11]质谱数据的比对,推测化合物23为雷公藤春碱。化合物31在正离子模式下产生m/z 858.285 4的准分子离子峰,二级质谱中产生脱去1分子H2O的m/z 840.275 7的碎片离子峰,准分子离子峰脱去1分子CH3COOH形成较强的m/z 798.263 8峰,在进一步脱去1分子CH3COOH形成738.243 5峰,准分子离子峰m/z 858.285 4脱去FuOH(C5H4O3)侧链形成的m/z 746.269 1的碎片离子峰,再进一步脱去1分子CH3COOH,形成m/z 686.248 0的碎片离子,m/z 206.082 5峰为大环开裂产生的吡啶二羧酸部分脱水产生的碎片离子,该离子进一步脱羧形成m/z 178.087 1的碎片离子,通过数据库和参考文献[20]质谱数据的比对,推测化合物31为雷公藤晋碱。雷公藤晋碱中吡啶二羧酸部分较雷公藤春碱中少一个羟基,故其易产生m/z 206.0825的碎片离子峰,并通过脱羧产生m/z 178.087 1峰。两种化合物的质谱图见图3。以雷公藤晋碱为例,解析此类化合物的裂解规律,见图4。因此得出吡啶二羧酸部分含有羟基的生物碱会产生m/z 204系列的特征碎片离子峰,不含羟基的生物碱则产生m/z 206系列的特征碎片离子峰。
3.2.4 萜类化合物结构解析
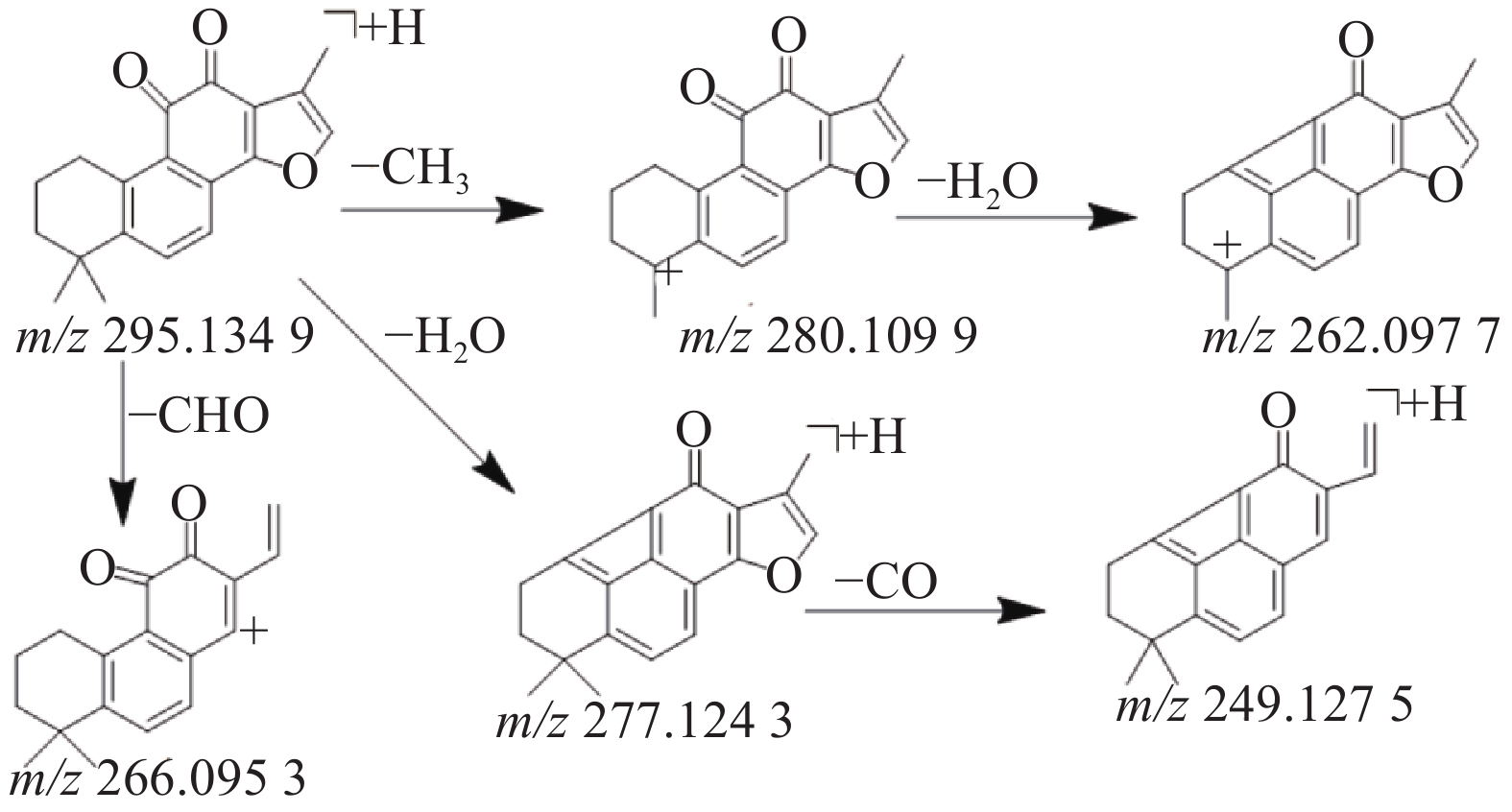
本研究共鉴定出8种萜类化合物,其中源于丹参药材中的5种萜类成分,丹参中的萜类化合物因其结构中主要含有羟基,羰基等取代基,所以质谱碰撞中主要丢失H2O,CO和-CH3等中性分子,产生一系列的碎片离子峰。化合物41在正离子模式下产生m/z 295.134 9的[M+H]+准分子离子峰,二级质谱中产生脱去1分子甲基形成的m/z 280.1099的碎片离子峰,在此基础上有丢失1分子水形成m/z 262.097 7峰,准分子离子峰脱去1分子H2O或脱去1个-CHO形成m/z 277.124 3峰或m/z 266.095 3峰,m/z 249.127 5峰是m/z 277.124 3脱去1分子H2O形成的碎片峰,通过对照品的保留时间和参考文献[10,18,23]数据比对,鉴定该化合物为丹参酮Ⅱ A,其质谱裂解规律见图5。
来源于雷公藤药材中的3种二萜类成分,该类化合物的二级质谱中出现一系列的脱水、脱CO和异丙基等碎片离子峰。化合物11在正离子模式下产生m/z 361.166 5的准分子离子峰,脱去2分子H2O和2分子CO形成m/z 269.154 3的碎片离子峰,m/z 227.108 3为m/z 269.154 3脱去1分子CH2CHCH3形成的碎片离子,其进一步脱1分子H2O和HCHO形成m/z 185.096 9的碎片离子,通过对照品比对和参考文献[14-15]的质谱数据,确定化合物10为雷公藤甲素。化合物18在正离子模式下产生m/z 359.148 9的准分子离子峰,脱去2分子H2O和2分子CO形成m/z 267.138 0的碎片离子峰,m/z 225.019 5为m/z 267.138 0脱去1分子CH2CHCH3形成的碎片离子,其进一步脱1分子H2O和HCHO形成m/z 183.0799的碎片离子,通过对照品比对和参考文献[19]的质谱数据,确定化合物16为雷公藤内酯酮。化合物39在负离子模式下产生m/z 299.199 6 的准分子离子峰,二级质谱中产生m/z 283.168 2的碎片离子, 提示为丢失1个-CH3后形成双键产生的碎片离子峰,A环发生RDA裂解产生m/z 213.090 8的碎片离子峰,通过数据库比对和参考文献[22]的质谱数据,推测化合物39为雷酚萜。
3.2.5 酚酸类化合物结构解析
在正负离子模式下共鉴定出乙酸乙酯部位中5种酚酸类成分,均来自于丹参药材,参考文献[18]报道的丹参中酚酸类成分的裂解规律发现,酚酸类化合物主要含有羰基、羧基和羟基,所以在质谱碰撞中易丢失CO、H2O和CO2的中性碎片;丹参素和咖啡酸作为基本母核而其他的水溶性酚酸类化合物大多数为这两者的聚合或缩合产物,主要为缩酚酸类的成分,在质谱碰撞中易丢失[M-H-180]−和[M-H-198]−中性碎片;含有羧基的单体化合物在负离子模式下会产生135[C8H7O2]−和179[C9H7O4]−的特征性碎片。化合物5中,在负离子模式下产生m/z 197.044 7的[M-H]−准分子离子峰,二级质谱进一步产生丢失1分子H2O和1分子CO2,形成的m/z 179.038 3和m/z 135.044 3的碎片离子峰,推测出结构中含有羧基,结合其精确分子量和参考文献[10]质谱数据,推测该化合物为丹参素。化合物12中,负离子模式下产生m/z 491.097 0的[M-H]−准分子离子峰,二级质谱中产生m/z 311.054 9和m/z 293.044 6的碎片离子峰,分别为[M-H-180]−和[M-H-198]−, m/z 267.064 6峰为m/z 311.054 9脱去1分子CO2所产生,根据m/z 135.044 7峰推测结构中含有羧基,结合其精确分子量和参考文献[18]质谱数据的比较,推测该化合物为丹酚酸C。化合物1中,正离子模式下给出m/z 139.039 4的[M+H]+准分子离子峰,脱去1分子H2O形成m/z 121.028 7的碎片离子峰,通过数据库比对和参考文献[10],推测该化合物1为原儿茶醛。化合物13中,在正离子模式下产生m/z 341.066 9的[M+H]+准分子离子峰,脱去1分子CO2形成m/z 295.060 7的碎片离子峰,m/z 277.050 9和m/z 249.056 0的碎片离子峰是m/z 295.060 7峰分别脱去1分子H2O和1分子CO2形成的,通过数据库比对和参考文献[17]质谱数据,推测该化合物为丹酚酸G。
4. 讨论
4.1 色谱与质谱条件考察
本实验流动相考察了乙腈-水系统和甲醇-水系统,结果乙腈-水系统中化合物的分离度较好,加入甲酸可以改善峰形,有助于化合物的离子化,提高质谱的响应,最终选择乙腈-0.1%甲酸水系统作为本次研究的流动相。
4.2 化学成分的定性分析
据以往文献中三色片各化学成分的研究报道,收集各药材的主要化学成分的精确分子量,碎片离子峰等信息,建立相应的化学成分数据库。通过数据库比对,对照品保留时间及参考文献中质谱数据鉴定三色片醇提物乙酸乙酯部位的化学成分。本研究共鉴定出42个化合物,其中5个是通过对照品鉴定得出,对无对照品的化合物,通过质谱的裂解特征及参考文献进行结构表征,对同分异构体应结合其在液相色谱中化合物的保留时间及质谱行为,综合对其定性鉴别。
三色片醇提物的乙酸乙酯部位具有较强的抗补体活性,本研究采用UPLC-Q-TOF-MS法对其中的化学成分进行结构表征,结果发现该部位主要含有生物碱类,萜类,黄酮和酚酸类等化学成分。其中以来源于雷公藤药材中极性中等的生物碱类成分含量较多,这与三色片提取物的制备工艺有关,三色片中雷公藤药材采用乙醇加热回流提取的方式,而黄芪和丹参药材采用水提取醇沉淀的方式。此外,先前的研究发现广藿香中的黄酮和萜类化合物对旁路途径的补体激活具有明显的抑制作用,紫花地丁中的生物碱类成分对旁路途径也有抑制作用(AP50=0.22~0.50 g/L), 牡丹皮和毛七公的抗补体活性成分研究中发现酚羟基决定抗补体活性的存在与否,没食子酰基可改善抗补体活性,甲基则对抗补体活性不利[25-27]。通过本次研究对三色片醇提物的乙酸乙酯部位的化学成分进行了初步表征,为阐明三色片的药效物质基础提供参考依据。研究的不足之处在于,仍有部分化学成分尚未定性鉴定,含量较高的单体成分未进行体外抗补体活性的测定,未来将通过中药化学的方法获得含量较高的单体成分,并进行结构鉴定和抗补体活性测定。
-
表 1 全长克隆引物
引物名称 引物序列 PRR1-F ACCTCAAGGGCCACTGGTTC PRR1-R GTAACAAAAGACTTTCTGAA 表 2 UHPLC-MS分析方法
色谱条件 质谱条件 色谱柱:Agilent ZORBAX C18(3.5 μm,2.1 mm ×100 mm) 毛细管电压:3.5 kV 流动相:A:0.1%甲酸-水,B:乙腈 锥孔电压:60 V 流速:0.4 ml/min 离子源温度:350 ℃ 柱温:40 ℃ 碎裂电压:120 V 进样量:0.4 ml 采集范围:50~1 100 m/z 梯度洗脱:0 min,5%B; 4 min,20%B;
6 min, 21% B; 9 min, 26% B; 11 min, 40% B; 15 min, 80% B; 17 min, 95% B表 3 酵母双杂筛选互作蛋白
序号 蛋白家族名称 预测功能 1 热休克蛋白 广泛参与植物响应外界环境胁迫,生物合成以及生长发育相关的转录调控 2 热休克蛋白 3 AP2转录因子 4 AP2转录因子 5 AP2转录因子 -
[1] 王升, 蒋待泉, 康传志, 等. 药用植物次生代谢在中药材生态种植中的作用及利用[J]. 中国中药杂志, 2020, 45(9):2002-2008. [2] DUNLAP J C. Molecular bases for circadian clocks[J]. Cell,1999,96(2):271-290. doi: 10.1016/S0092-8674(00)80566-8 [3] BENDIX C, MARSHALL C M, HARMON F G. Circadian clock genes universally control key agricultural traits[J]. Mol Plant,2015,8(8):1135-1152. doi: 10.1016/j.molp.2015.03.003 [4] 徐小冬, 谢启光. 植物生物钟研究的历史回顾与最新进展[J]. 自然杂志, 2013, 35(2):118-126. [5] BECKWITH E J, YANOVSKY M J. Circadian regulation of gene expression: at the crossroads of transcriptional and post-transcriptional regulatory networks[J]. Curr Opin Genet Dev,2014,27:35-42. doi: 10.1016/j.gde.2014.03.007 [6] ITO S, MATSUSHIKA A, YAMADA H, et al. Characterization of the APRR9 pseudo-response regulator belonging to the APRR1/TOC1 quintet in Arabidopsis thaliana[J]. Plant Cell Physiol,2003,44(11):1237-1245. doi: 10.1093/pcp/pcg136 [7] COCKRAM J, THIEL T, STEUERNAGEL B, et al. Genome dynamics explain the evolution of flowering time CCT domain gene families in the Poaceae[J]. PLoS One,2012,7(9):e45307. doi: 10.1371/journal.pone.0045307 [8] 陈华夏, 申国境, 王磊, 等. 4个物种CCT结构域基因家族的序列进化分析[J]. 华中农业大学学报, 2010, 29(6):669-676. [9] MATSUSHIKA A, MAKINO S, KOJIMA M, et al. Circadian waves of expression of the APRR1/TOC1 family of pseudo-response regulators in Arabidopsis thaliana: insight into the plant circadian clock[J]. Plant Cell Physiol,2000,41(9):1002-1012. doi: 10.1093/pcp/pcd043 [10] KURUP S, JONES H D, HOLDSWORTH M J. Interactions of the developmental regulator ABI3 with proteins identified from developing Arabidopsis seeds[J]. Plant J,2000,21(2):143-155. doi: 10.1046/j.1365-313x.2000.00663.x [11] LEGNAIOLI T, CUEVAS J, MAS P. TOC1 functions as a molecular switch connecting the circadian clock with plant responses to drought[J]. EMBO J,2009,28(23):3745-3757. doi: 10.1038/emboj.2009.297 [12] GRUNDY J, STOKER C, CARRÉ I A. Circadian regulation of abiotic stress tolerance in plants[J]. Front Plant Sci,2015,6:648. [13] SEO P J, MAS P. STRESSing the role of the plant circadian clock[J]. Trends Plant Sci,2015,20(4):230-237. doi: 10.1016/j.tplants.2015.01.001 [14] SALOMÉ P A, WEIGEL D, MCCLUNG C R. The role of the Arabidopsis morning loop components CCA1, LHY, PRR7, and PRR9 in temperature compensation[J]. Plant Cell,2010,22(11):3650-3661. doi: 10.1105/tpc.110.079087 [15] NAKAMICHI N, KIBA T, HENRIQUES R, et al. PSEUDO-RESPONSE REGULATORS 9, 7, and 5 are transcriptional repressors in the Arabidopsis circadian clock[J]. Plant Cell,2010,22(3):594-605. doi: 10.1105/tpc.109.072892 [16] MIZUNO T. Two-component phosphorelay signal transduction systems in plants: from hormone responses to circadian rhythms[J]. Biosci Biotechnol Biochem,2005,69(12):2263-2276. doi: 10.1271/bbb.69.2263 [17] 王晗. 生物钟生物学及其研究进展[J]. 生命科学, 2015, 27(11):1313-1319. [18] 杨志福, 梅其炳, 蒋永培. 红花有效成分及药理作用[J]. 西北药学杂志, 2001, 16(3):131-133. doi: 10.3969/j.issn.1004-2407.2001.03.025 [19] 刘飞. 红花黄酮类化合物生物合成途径关键酶基因的克隆与功能验证[D]. 上海: 第二军医大学, 2014. [20] GUO D D, GAO Y, LIU F, et al. Integrating molecular characterization and metabolites profile revealed CtCHI1& apos; s significant role in Carthamus tinctorius L[J]. BMC Plant Biol,2019,19(1):1-13. doi: 10.1186/s12870-018-1600-2 [21] BROWN B A, JENKINS G I. UV-B signaling pathways with different fluence-rate response profiles are distinguished in mature Arabidopsis leaf tissue by requirement for UVR8, HY5, and HYH[J]. Plant Physiol,2008,146(2):576-588. [22] FARRÉ E M, WEISE S E. The interactions between the circadian clock and primary metabolism[J]. Curr Opin Plant Biol,2012,15(3):293-300. doi: 10.1016/j.pbi.2012.01.013 [23] 何贝轩, 郭丹丹, 贾鑫磊, 等. 昼夜节律钟调控花青素类成分的生物合成[J]. 药学学报, 2018, 53(3):345-355. 期刊类型引用(1)
1. 齐书艺,王璐暖,何贝轩,高越,郭美丽. 红花黄酮生物合成途径糖基转移酶基因CtUGT25的功能研究. 药学学报. 2024(06): 1854-1863 .  百度学术
百度学术其他类型引用(3)
-




 下载:
下载:
 下载:
下载: